ESPRI est un centre de données du Pôle National Atmosphère AERIS
.................................
Accueil ACOMIDA
......................................
Groupe de Travail ADOMOCA
......................................
Lettre ADOMOCA
......................................
Maquette ADOMOCA
.................................
Maquette ADOMOCA (Version 4) : Assimilation DOnnées dans des MOdèles de Chimie Atmosphérique
- Présentation du projet ADOMOCA
Ce projet a pour but d'organiser au mieux la communauté chimie atmosphérique française autour
de l'assimilation des données chimiques troposphériques et stratosphériques existantes et à venir.
L'éventail des outils disponibles (modèles, mesures spatiales, méthodes d'assimilation) a favorisé
l'utilisation du coupleur PALM développé au CERFACS. Deux modèles ont été sélectionnés :
MOCAGE au CNRM et LMDz-INCA à l'IPSL (en commençant par l'outil MSDOL). Deux ingénieurs en CDD (CERFACS et IPSL)
ont été financés par le CNES pour PALMériser les modèles et les méthodes d'assimilation
(3DFGAT, 4DVAR et interpolation linéaire), et aider la communauté scientifique à développer
les opérateurs d'observation pour les différents senseurs spatiaux.
Les outils nécessaires (fichiers de forcage, sources, scripts) à la mise en oeuvre de la chaïne PALMée MOCAGE sont fournis ci-dessous.
- La documentation ADOMOCA
- Une documentation exhautive sur la maquette ADOMOCA est disponible : cliquer ici.
- Les fichiers ADOMOCA
Pour récupérer les fichiers de la maquette ADOMOCA : cliquer sur le fichier en utilisant la commande --> save link as- ADOMOCA_V4.tgz (344 Ko) : sources des routines d'assimilation correspondantes aux unités PALM et des interfaces entre modèles et coupleur PALM.
- MOCAGE_CONSTANTS.tgz (43 Mo) : fichiers de constantes pour les réactions de photolyse et pour le schéma d'ozone linéaire.
- MOCAGE_DATA_47NIV.tgz (332 Mo) : fichiers d'entrée (pour la date du 1 juillet 2003) pour la version avec 47 niveaux sur la verticale (forçages dynamiques issus d'Arpège).
- MOCAGE_DATA_60NIV.tgz (633 Mo) : fichiers d'entrée (pour la date du 1 juillet 2003) pour la version avec 60 niveaux sur la verticale (forçages dynamiques issus du modèle de l'ECMWF).
- ADOMOCA_EXT_LIBS.tgz (2.5 Mo) : sources et les instructions pour compiler les librairies FA/LFI, GRIBEX, NOVELTIS, SPARSKIT.
- ADOMOCA_VISU.tgz (2.5 Mo) : "go-files" ferret pour la visualisation des fichiers NetCDF et les fichiers NetCDF d'orographie globale pour le traçage en coordonnée vert. km.
- MOCAGE_DIRECT_POUR_V4.tgz (381 Ko) : contient les sources du modèle direct avce les schémas linéaires pour O3, CO, HNO3 et traceur froid (en attente d'une release officielle par le CNRM)
La documentation ADOMOCA (télécharger le fichier pdf)
Plan du document :
Introduction
Les différences principales par rapport à la version 3 résident dans : l'évolution du schéma linéaire - qui peut maintenant modéliser O3, CO, HNO3 et traceur froid - ; dans la possibilité de remplacer le modèle par un chargeur de sorties NetCDF - pour la répétition rapide de cas d'assimilation ou pour la comparaison à posteriori avec de nouveaux jeux d'observations - ; l'amélioration de l'assimilation des colonnes totales - par reconstruction d'un profil vertical équivalent - ; l'optimisation de la mémoire utilisée par les opérateurs d'observation.
La constante évolution de la représentation de B - calculs d'assimilation effectuées sur une grille de Gauss optimale pour la solution spectrale de l'équation de diffusion, la refonte de l'approximation de la corrélation verticale, l'utilisation de variances et de longuers de portée diagnostiquées - et l'introduction d'un seuil sur la valeur minimale admissible pour les observations et les analyses ont permis d'améliorer les performances de l'assimilation.
Les scripts de lancement ont été adaptés au nouveaux schémas linéaires et au chargeur NetCDF.
La maquette est basée sur le modèle global de chimie transport MOCAGE (avec 4 schémas chimiques différents, grille rectangulaire 2º x 2º ou 0,5º x 0,5º ou de Gauss en troncature triangulaire T42 ou T21 et deux résolutions verticales) et sur le modèle global 2.5º x 2.5º de chimie transport MSDOL. Avec la nouvelle politique de diffusion les sources des modèles ne sont plus distribuées avec la maquette : la refonte du système de compilation (Makefile et scripts d'installation des sources) garantit la pleine compatibilité entre la compilation du modèle direct et celle des routines d'assimilation.
La maquette implémente la méthode variationnelle dite 3D-FGAT (3D VAR with First Guess at Appropriate Time) implémentée grâce au coupleur dynamique PALM.
Ce document n'est pas un manuel du modèle MOCAGE, ni du modèle MSDOL ni du coupleur PALM et encore moins une introduction à la méthode 3D-FGAT.
Il ne contient que la description des fichiers fournis et les instructions pour les installer et pour faire tourner des cas test.
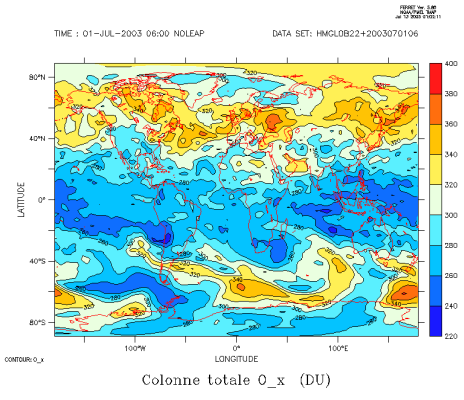
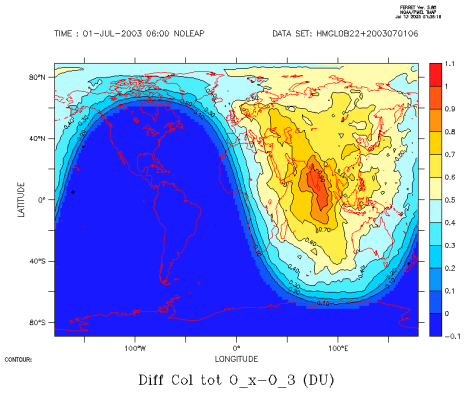
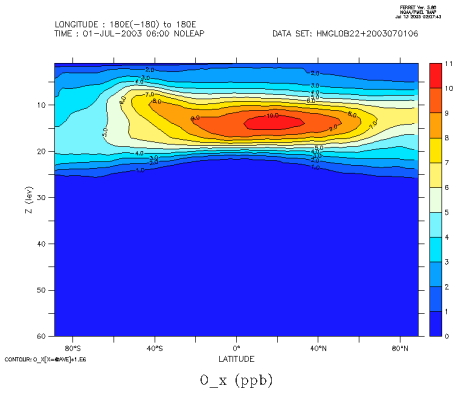
Ces cas concernent la journée du 1 juillet 2003 avec assimilation de profils satellitaires d'ozone, de type MIPAS.
En ce qui concerne le modèle, nous nous limitons à rappeler que MOCAGE est un code de chimie transport modulaire, qui permet de choisir le schéma chimique à utiliser parmi une large gamme d'alternatives. Nous n'en avons retenu que 4 pour la maquette ADOMOCA :
- Le schéma CARIOLLE : il s'agit d'un schéma linéaire avec de une à quatre espèces (O3 stratosphérique v2.8, CO, HNO3 et traceur froid v1.2). Très stable et léger, le schéma CARIOLLE est le schéma idéal pour les tests et la mise au point.
- Le schéma REPROBUS : il s'agit d'un schéma avec 38 espèces transportées et 16 espèces à courte durée de vie adapté à la stratosphère. Schéma intermédiaire, encore relativement léger, mais avec une réelle prise en compte de la chimie. Permet de vérifier que l'algorithme d'assimilation sera robuste.
- Le schéma RACMOBUS : associe à REPROBUS le schéma RACM troposphérique. Avec ses 89 espèces transportées et 29 à courte durée de vie, c'est un schéma complet, mais gourmand en ressources de calcul.
- Le schéma RELACS : il s'agit d'un schéma
tropo-stratosphérique simplifié adapté aux
simulations climatiques longues. Avec ses 61 espèces
transportées
et 17 à courte durée de vie, la possibilité
de tourner sur une grille de Gauss 128 x 64 (clé CPP GAUSS) et
la simplification du traitement de la couche limite (clé CPP
SBL) avec contrainte sur la conservations de la masse (clé CPP
MASSy) c'est un schéma complet mais encore
suffisamment léger pour tourner sur de petites plates-formes.
- domaine global avec grille horizontale régulière 2º x 2º (180 x 90 mailles) et 47 niveaux hybrides sigma sur la verticale. Pour cette configuration, les forçages dynamiques sont issus de simulations Arpège à 41 niveaux (interpolation verticale en pression de type exponentiel)
- domaine global avec grille horizontale régulière 2º x 2º (180 x 90 mailles) et 60 niveaux hybrides sigma sur la verticale. Pour cette configuration, les forçages dynamiques sont issus de simulations du modèle IFS du CEPMMT à 60 niveaux (interpolation verticale en pression de type demi-somme)
- domaine global avec grille horizontale de Gauss correspondante à une troncature triangulaire T42 (128 x 64 mailles) et 60 niveaux hybrides sigma sur la verticale. Pour cette configuration, les forçages dynamiques sont issus de simulations du modèle IFS du CEPMMT à 60 niveaux (interpolation verticale en pression de type demi-somme)
- domaine global avec grille horizontale régulière 0.5º x 0.5º (720 x 360 mailles) et 47 niveaux hybrides sigma sur la verticale. Pour cette configuration, les forçages dynamiques sont issus de simulations Arpège à 41 niveaux (interpolation verticale en pression de type exponentiel)
- domaine global avec grille horizontale régulière 0.5º x 0.5º (720 x 360 mailles) et 60 niveaux hybrides sigma sur la verticale. Pour cette configuration, les forçages dynamiques sont issus de simulations du modèle IFS du CEPMMT à 91 niveaux (interpolation verticale en pression de type demi-somme)
Pour ce qui est des environnements de compilation et de production, nous avons opté pour trois plates-formes représentatives :
- le NEC SX8r de Météo - France avec MPI
propriétaire
pour le message passing et OpenMP
pour la parallélisation du
modèle
- un PC linux avec compilateur Portland Group pgf90 et LAM MPI pour le message passing
- l'Opteron bi-processeur avec compilateur Portland Group pgf90 et LAM MPI pour le message passing en service au Laboratoire d'Aérologie (aerosv1)
d'autres plates-formes de calcul.
Les fichiers de sortie sont au format FA/LFI (pour les fichiers de restart) et au format auto descriptif NetCDF (convention CF) pour les diagnostiques et la visualisation.
La maquette utilise donc les librairies XRD, GRIB et NetCDF. La troisième étant distribuée sur le web et très facile à installer, nous ne fournirons que les fichiers sources et les instructions de compilation pour les deux premières : à terme une version à jour et portable de ces deux bibliothèques sera distribuée$ et documentées avec les sources du modèle direct et remplacera la version distribuée avec la maquette.
L'opérateur d'observation est basé sur les routines d'interpolation développées par NOVELTIS qui sont installées séparément sous forme de librairie et qui s'appuient à leur tour sur la librairie d'algèbre linéaire pour matrices creuses SPARSKIT.
La modélisation de la matrice B est basée sur des routines des librairies open-source SPHEREPACK et SCRIP, directement inclues dans les source de la chaîne et sur la librairie d'algèbre linéaire LAPACK, qui est couramnnent installée sur toute les plates-formes unix/linux.
PALM s'appuie, pour la partie communication sur le standard de message passing MPI1.2 et pour la partie algébrique sur la librairie d'algèbre BLAS.
Nous supposons que les librairies et les outils correspondants sont déjà installés. Dans le cas contraire, des distributions libres de ces deux produits existent (e. g. LAM MPI ou mpich et l'implémentation optimisée BLAS de Kazushige Goto).
L'interface graphique PrePALM est codée en Tcl/Tk. Vous trouvez plus de détail sur les bibliothèques requises par PALM et PrePALM dans la documentation sur l'installation de PALM_RESEARCH.
Pour finir, nous présentons quelques outils pour la visualisation et l'exploration des résultats au format NetCDF. En particulier nous utiliserons le logiciel de visualisation Ferret.
Les ingrédients de la maquette
ADOMOCA_V4.tgzcontient les sources des routines d'assimilation correspondantes aux unités PALM et des interfaces entre modèles et coupleur PALM.
MOCAGE_CONSTANTS.tgzcontient des fichiers de constantes pour les réactions de photolyse et pour les schémas linéaires
MOCAGE_DATA_47NIV.tgzcontient les fichiers d'entrée (pour la date du 1 juillet 2003) pour la version avec 47 niveaux sur la verticale (forçages dynamiques issus d'Arpège)
MOCAGE_DATA_60NIV.tgzcontient les fichiers d'entrée (pour la date du 1 juillet 2003) pour la version avec 60 niveaux sur la verticale (forçages dynamiques issus du modèle de l'ECMWF)ADOMOCA_EXT_LIBS.tgzcontient les sources et les instructions pour compiler les librairies FA/LFI, GRIBEX, NOVELTIS, SPARSKIT
ADOMOCA_VISU.tgzcontient des "go-files" ferret pour la visualisation des fichiers NetCDF et les fichiers NetCDF d'orographie globale pour le traçage en coordonnée vert. km.MOCAGE_DIRECT_POUR_V4.tgzcontient les sources du modèle direct avce les schémas linéaires pour O3, CO, HNO3 et traceur froid (en attente d'une release officielle par le CNRM)
N.B. Le format tgz correspond à des archives
tar compressées avec gzip.Pour en visualiser le contenu:
tar tvzf
XXXX.tgztar xvzf
XXXX.tgzInstallons les EXT_LIBS...
Si vraiment il faut le faire, autant le faire tout de suite!Ces librairies sont déjà installées sur les machine de Météo - France et les Makefile fournis avec la maquette pointent au bon emplacement.
Pour les autres machines il faut les extraire de l'archive
ADOMOCA_EXT_LIBS.tgz
et les compiler.Dans un répertoire de votre gré, extrayez le contenu de
ADOMOCA_EXT_LIBS.tgztar xvzf ADOMOCA_EXT_LIBS.tgzVous obtiendrez un fichier et trois nouvelles archives compressées.
facadi.F
gribex_000263.tar.gz
xrd.benchmark.tar.gz
noveltis+sparskit.tgz
Installation de GRIBEX
Il faut extraire l'archive compresséetar xvzf gribex_000263.tar.gzLes instructions qui suivent correspondent à un environnement linux avec compilateurs Portland Group, mais elles s'appliquent à peu de choses près à d'autres environnements linux.
Dans le répertoire
gribex_000263 exécuter
le script build_libraryChoisir les compilateurs pgf90 et la promotion des réels à 64 bit
A la fin de la première compilation répondre que l'on ne veut pas de bibliothèque read only.
Modifier le fichier
config/config.linuxR64 pour
insérer les options -pc 64 -byteswapio -O3 -MdalignTaper
make cleanet enfin
makePour d'autres plates-formes le script build_library pourrait ne pas marcher, mais les instructions contenues dans le package vont indiquer les fichiers à modifier à la main pour compiler.
A la fin de la compilation vous disposerez de la librairie
libgribexR64.a
dont vous aurez besoin pour l'édition de liens de MOCAGE.Installation de FA/LFI
La deuxième librairie qu'il faut installer est un sous-ensemble de la librairie XRD de Météo - France, et en particulier celle qui concerne les entrées/sorties au format FA/LFI. Cependant une différence dans la représentation de la coordonne verticale hybride nous oblige à remplacer un fichier de la distribution par une version modifiée (facadi.F).Il faut extraire l'archive compressée
tar xvzf xrd.benchmark.tar.gzDans le répertoire
xrd:remplacer le fichier
fa/facadi.F par le fichier facadi.F
qui se trouve dans MOCAGE_EXT_LIBS.tgzEnsuite exécuter dans l'ordre
cd fa
\rm *.o *.mod
pgf90 -r8 -pc 64 -byteswapio -O3 -Mdalign -I../include -DLITTLE_ENDIAN
-c famodu.F
pgf90 -r8 -pc 64 -byteswapio -O3 -Mdalign -I../include -DLITTLE_ENDIAN
-c *.F
cd ../lfi
\rm *.o *.mod
pgf90 -r8 -pc 64 -byteswapio -O3 -Mdalign -I../include -DLITTLE_ENDIAN
-c *.F
cd ../grib_mf
\rm *.o *.mod
pgf90 -r8 -pc 64 -byteswapio -O3 -Mdalign -I../include -DLITTLE_ENDIAN
-c *.F
cd ../not_used
\rm *.o *.mod
pgf90 -r8 -pc 64 -byteswapio -O3 -Mdalign -I../include -DLITTLE_ENDIAN
-c is*.F
pgf90 -r8 -pc 64 -byteswapio -O3 -Mdalign -I../include -DLITTLE_ENDIAN
-c abor2.F
cd ..
ar ruv libxrd_jfe.a fa/*.o lfi/*.o grib_mf/*.o not_used/*.o
Bien sur, la commande
pgf90 -r8 -pc 64 -byteswapio -O3
-Mdalign se réfère à l'environnement
Portland Group sur
PC linux et elle doit être remplacée par la commande de
compilation qui a été employée pour gribex. Faites
attention à ne pas oublier la promotion des réels
(l'équivalent de l'option -r8).Le nom
libxrd_jfe.a a été attribué
en hommage à Jean François Estrade qui s'est
démené pour nous procurer une version portable de FA/LFI.Installation des
bibliothèques NOVELTIS
et SPARSKIT
Les librairies qui sont à la base de l'opérateur
d'observation sont distribuées dans une seule archive.Il faut extraire l'archive compressée
tar xvzf noveltis+sparskit.tgzDans le répertoire
source du répertoire NOVELTIS
on trouve des modèles de fichier makefile.inc.xxx Il faut
éditer celui qui correspond à la configuration de votre
machine et le renommer ou l'associer par lien au nom makefile.incEnsuite il suffit de lancer la compilation par
makeDans le répertoire
SPARSKIT il faut éditer
les premières lignes du makefile pour choisir les
commandes et les options de compilations compatibles avec les choix
effectués pour la librairie Noveltis.Ensuite il suffit de lancer la compilation par
makeOuvrons les gros
morceaux...
Pas de peur! Les plus gros fichiers sont les moins méchants. Il
s'agit des données d'entrée pour le modèle direct
et pour la chaîne d'assimilation.Il y a deux versions puisque nous gérons deux résolutions verticales différentes pour la grille 2° x 2° et pour des observations de type MIPAS (profils sans averaging kernel). Les fichiers pour la grille de Gauss
GLOT42 sont fournis pour
les schémas RELACS et CARIOLLE, exclusivement pour la
discrétisation verticale à 60 niveaux. Des exemples pour
d'autres
types d'observations sont disponibles sur demande.Les données contenues dans ces archives ne servent que pour l'exécution (pas pour la compilation) et peuvent donc être extraites directement dans le répertoire où tournera l'application ou dans un espace de stockage accessible depuis la machine de production. Dans le cas de Météo France elles sont stockées sur la machine d'archivage
cougar (alias
delage) sous le chemin../../mrgm/mrgm205/MOCAGE_V1/DATA/47NIVou
../../mrgm/mrgm205/MOCAGE_V1/DATA/60NIVVoyons ce qu'il y a dedans :
MOCAGE_DATA_47NIV.tgz
Données
d'entrée pour la configuration à 47 niveaux domaine
global, résolution horizontale 2° x 2°, pour la
journée du 1er juillet 2003 :47NIV/HM/HMGLOB22+200307010047NIV/FM/FMGLOB22+2003070100
47NIV/FM/FMGLOB22+2003070103
47NIV/FM/FMGLOB22+2003070106
47NIV/FM/FMGLOB22+2003070109
47NIV/FM/FMGLOB22+2003070112
47NIV/FM/FMGLOB22+2003070115
47NIV/FM/FMGLOB22+2003070118
47NIV/FM/FMGLOB22+2003070121
47NIV/FM/FMGLOB22+200307020047NIV/SURF/[RACMOBUS, RELACS]_SURF47NIV/SM/SMGLOB22+20030701_[CARIOLLE,
REPROBUS, RACMOBUS]
47NIV/CH/Jdata07.bin47NIV/OBSERVATIONS/HOBS+20030701
47NIV/ASSIM/HDAT+20030701
47NIV/ASSIM/HCOV+20030701HDAT,
au format MOCAGE (ascii).MOCAGE_DATA_60NIV.tgz
Données
d'entrée pour la configuration à 60 niveaux domaine
global, résolution
horizontale 2° x 2°, pour la journée du 1er juillet 2003
:60NIV/HM/HMGLOB22+200307010060NIV/HM/HMGLOT42+200307010060NIV/FM/FMGLOB22+2003070100
60NIV/FM/FMGLOB22+2003070106
60NIV/FM/FMGLOB22+2003070112
60NIV/FM/FMGLOB22+2003070118
60NIV/FM/FMGLOB22+2003070200
60NIV/FM/FMGLOT42+2003070100
60NIV/FM/FMGLOT42+2003070106
60NIV/FM/FMGLOT42+2003070112
60NIV/FM/FMGLOT42+2003070118
60NIV/FM/FMGLOT42+200307020060NIV/SURF/[RACMOBUS, RELACS]_SURF60NIV/SM/SMGLOB22+20030701_[CARIOLLE,
RELACS, REPROBUS, RACMOBUS]
60NIV/SM/SMGLOT42+20030701_RELACS
60NIV/CH/Jdata07.bin
60NIV/OBSERVATIONS/HOBS+20030701
60NIV/ASSIM/HDAT+2003070160NIV/ASSIM/HCOV+20030701HDAT,
au format MOCAGE (ascii).Le format des
fichiers d'observations HOBS, HDAT, de covariance
des
erreurs d'observation HCOV et d'averaging kernel HAVK
Les observations sont stockées dans des fichiers ASCII au
format d'observation MOCAGE, aussi dit HOBS ou HDAT.
Les deux types de fichiers ont le même format, mais les HOBS
contiennent des observations pour vérification et sont
utilisés lors de l'intégration du modèle direct,
tandis que les HDAT contiennent les données
à assimiler et sont utilisés par le 3DFGAT. Normalement
un fichier est créé par date.Le format prévoit une entête de deux entiers avec le nombre total de profils (il faut comprendre points de mesure sur le plan lon, lat : une colonne totale est comptée comme un profil dans le sens qu'elle correspond à une position géographique) et le nombre de types d'observations (c'est à dire le nombre d'instruments). Par exemple une première ligne
1203 1signifie que le fichier décrit 1203 observations d'un seul type.
Ensuite pour chaque type une ligne (chaîne de caractères) identifie le type. Par exemple
MIPAS OZONE PROFILESi le fichier contient plusieurs types d'observations chaque ligne doit indiquer le nombre de profils par instrument avec le mot clé
#P=.
Par
exemple la ligneMIPAS OZONE PROFILE #P=846signifie que le le type
MIPAS OZONE PROFILE contient 846
profils. Le mot clé
TOTCOL TOMS OZONE TOTCOLLe mot clé
AVKERN indique que le profil utilise
une fonction de balayage (ou averaging
kernel). Dans ce cas les matrices d'averaging kernel seront
stockées dans des fichiers HAVK. Par exempleMOPITT CO PROFILE ( AVKERN accounted )
Le mot clé INTQTY indique une
quantité intégrée scalaire obtenue par application
d'une fonction de balayage (ou averaging
kernel) à un profil vertical intermédiaire. Le
nombre de niveaux du profil intermédiaire doit être le
même pour tous les points de mesure et il doit être
indiqué avec le mot clé : INTQTY=7. Dans ce
cas les matrices d'averaging kernel se réduisent à des
vecteurs
ligne et sont
également stockées dans les fichiers HAVK.
Par exempleMOPITT CO INTQTY=7 Enfin, il est possible de préciser si la covariance d'erreur d'observation pour ce type d'instrument doit être lue dans un fichier
HCOV (cas par default) ou si elle doit
être
calculée comme étant diagonale avec variances
calculées à partir d'un r.m.s. proportionnel à la
valeur de l'observation et exprimé en pourcentage. Dans ce cas
le mot clé %R= est utilisé. Par exempleMOPITT CO PROFILE ( AVKERN accounted ) %R=10.5Ensuite par profil on donne la date (à la minute près, au format
yyyymmddhhmm), deux chaînes de
caractères pour identifier l'instrument, la longitude, la
latitude et le nombre de niveaux. Par exemple200307010001 MIPAS 00000 -22.51 -23.59 8Remarque : pour le cas des mesures scalaires
INTQTY, le
nombre de niveaux doit être positionné à 1 (il
correspond au nombre de valeurs significatives, donc 1, scalaire, et
non pas au nombre de niveaux intermédiaires auxquels on applique
le averaging kernel.Pour chaque niveau, dans le cas de vrais profils deux entiers donnent la pression (en Pa) et le nombre d'espèces. Par exemple
14704 1Dans le cas de quantités intégrées
INTQTY,
on fera apparaitre autant de lignes que de niveaux sur le profil
intermédiaire, avec les valeurs en pression des niveaux.Dans le cas de colonnes totales
TOTCOL, deux entiers
donnent la pression (en Pa) des
bornes de la bande d'intégration (il est conseillé de
mettre 0 et 150000 pour les colonnes
totales) et un troisième entier le
nombre d'espèces. Par exemple 14704 28012 1Et ensuite, pour chaque espèce, l'identifiant de l'espèce et la valeur (en ppbv). Par exemple
[ O3] 7.845e-08Dans le cas de quantités intégrées
INTQTY,
seulement la première valeur par profil est significative. Les
valeurs des autres niveaux doivent apparaître pour
cohérence, mais elles sont ignorées.Et ainsi de suite.
Voilà un exemple avec la description de deux profils d'ozone MIPAS
1203 1
MIPAS OZONE PROFILE
200307010001 MIPAS 00000 -22.51 -23.59 8
14704 1
[ O3] 7.845e-08
8705 1
[ O3] 3.7895e-07
5314 1
[ O3] 1.50057e-06
3304 1
[ O3] 4.28395e-06
2108 1
[ O3] 6.5986e-06
1349 1
[ O3] 8.74225e-06
871 1
[ O3] 9.2004e-06
558 1
[ O3] 7.511e-06
200307010002 MIPAS 00000 -17.74 -24.85 8
14634 1
[ O3] 9.127e-08
8809 1
[ O3] 1.5001e-07
5392 1
[ O3] 1.33555e-06
3418 1
[ O3] 3.9125e-06
2126 1
[ O3] 6.45492e-06
1392 1
[ O3] 8.90177e-06
908 1
[ O3] 8.97024e-06
586 1
[ O3] 7.74211e-06Pour l'assimilation on associe à chaque observation l'estimation de la variance/covariance des erreurs associées. Cette information est stockée sous forme de matrice triangulaire dans un fichier
ASCII dont le nom commence par HCOV et
dont le format reprend celui des fichiers d'observations.La seule différence réside dans le fait qu'à chaque niveau, à la place du nombre d'espèces observées on indique le nombre des niveaux sous-jacents plus un (pour le niveau lui-même) c'est à dire le nombre d'entrées sur la ligne correspondante de la matrice triangulaire. A la place du nom de l'espèce on indique les deux indices de l'élément dans la matrice triangulaire.
Les deux triangles associés aux deux profils de l'exemple du fichier
HDAT sont stockés de la façon
suivante :1203 1
MIPAS OZONE PROFILE COVARIANCE
200307010001 MIPAS 00000 -22.51 -23.59 8
14704 1
[ 1, 1] 2.026269e+15
8705 2
[ 2, 1] 1.082391e+15
[ 2, 2] 8.555523e+14
5314 3
[ 3, 1] 3.991306e+14
[ 3, 2] 4.187799e+14
[ 3, 3] 3.873538e+14
3304 4
[ 4, 1] 1.947983e+14
[ 4, 2] 1.940480e+14
[ 4, 3] 2.093174e+14
[ 4, 4] 1.805103e+14
2108 5
[ 5, 1] 1.038566e+14
[ 5, 2] 1.022465e+14
[ 5, 3] 1.024486e+14
[ 5, 4] 1.028033e+14
[ 5, 5] 9.791682e+13
1349 6
[ 6, 1] 5.065115e+13
[ 6, 2] 5.117722e+13
[ 6, 3] 5.068160e+13
[ 6, 4] 4.670212e+13
[ 6, 5] 4.324100e+13
[ 6, 6] 5.176705e+13
871 7
[ 7, 1] 2.471180e+13
[ 7, 2] 2.548203e+13
[ 7, 3] 2.522136e+13
[ 7, 4] 2.215843e+13
[ 7, 5] 1.321102e+13
[ 7, 6] 2.632436e+13
[ 7, 7] 3.843973e+13
558 8
[ 8, 1] 1.771811e+13
[ 8, 2] 1.821650e+13
[ 8, 3] 1.824655e+13
[ 8, 4] 1.589768e+13
[ 8, 5] 8.373302e+12
[ 8, 6] 1.124872e+13
[ 8, 7] 2.368352e+13
[ 8, 8] 3.811663e+13
200307010002 MIPAS 00000 -17.74 -24.85 8
14634 1
[ 1, 1] 1.768776e+15
8809 2
[ 2, 1] 9.206902e+14
[ 2, 2] 6.755044e+14
5392 3
[ 3, 1] 3.613231e+14
[ 3, 2] 3.469656e+14
[ 3, 3] 3.544705e+14
3418 4
[ 4, 1] 1.703103e+14
[ 4, 2] 1.574354e+14
[ 4, 3] 1.878993e+14
[ 4, 4] 1.546838e+14
2126 5
[ 5, 1] 9.025739e+13
[ 5, 2] 8.435077e+13
[ 5, 3] 9.751882e+13
[ 5, 4] 9.275953e+13
[ 5, 5] 8.494587e+13
1392 6
[ 6, 1] 5.115158e+13
[ 6, 2] 4.743110e+13
[ 6, 3] 5.244395e+13
[ 6, 4] 4.561432e+13
[ 6, 5] 4.737985e+13
[ 6, 6] 5.202469e+13
908 7
[ 7, 1] 3.182392e+13
[ 7, 2] 2.890106e+13
[ 7, 3] 3.040286e+13
[ 7, 4] 2.501512e+13
[ 7, 5] 2.350402e+13
[ 7, 6] 2.524424e+13
[ 7, 7] 3.307055e+13
586 8
[ 8, 1] 2.092612e+13
[ 8, 2] 1.900108e+13
[ 8, 3] 2.017338e+13
[ 8, 4] 1.638635e+13
[ 8, 5] 1.462928e+13
[ 8, 6] 9.195132e+12
[ 8, 7] 1.814148e+13
[ 8, 8] 3.075253e+13Pour la description de profils avec averaging kernel, la valeur observée xo est liée à l'état modèle interpolé au point d'observation xm par la relation xo = xa + A (xm - xa) où x
a est un profil à priori et A est une
matrice (une par profil) de lissage dite averaging kernel. Puisque pour
l'assimilation nous sommes intéressés à la
différence entre valeur observée et valeur
estimée, il est plus économique de stocker dans le
fichier HDAT la quantité xo - (I-A)
xa de façon à la comparer directement
à la quantité Axm
produite par l'opérateur d'observation. La matrice A pour chaque profil est
stockée sous forme de matrice rectangulaire dense dans
un fichier ASCII dont le nom commence par HAVK et
dont le format reprend celui des fichiers HCOV.La seule différence réside dans le fait que, la matrice étant non symétrique, à chaque niveau, à la place du nombre d'espèces observées on indique le nombre des niveaux du profil c'est à dire le nombre d'entrées sur la ligne correspondante de la matrice A. A la place du nom de l'espèce on indique les deux indices de l'élément dans la matrice.
La matrice associée au premier profil d'un fichier
HDAT relatif à un profil MOPITT avec 7
niveaux sur la verticale est
stockée dans le fichier HAVK de la façon
suivante :
6214 1
MOPITT CO Averaging Kernel ( AVKERN )
200307010012 MOPITT 00000 -36.49 -18.73 7
101878 7
[ 1, 1] 8.32152E-02
[ 1, 2] 3.31613E-01
[ 1, 3] 5.02991E-01
[ 1, 4] 4.45117E-01
[ 1, 5] 2.08479E-01
[ 1, 6] 7.13190E-02
[ 1, 7] 2.74929E-02
85000 7
[ 2, 1] 7.22599E-02
[ 2, 2] 2.89049E-01
[ 2, 3] 4.46514E-01
[ 2, 4] 4.13668E-01
[ 2, 5] 2.19529E-01
[ 2, 6] 1.00776E-01
[ 2, 7] 6.27162E-02
70000 7
[ 3, 1] 5.15932E-02
[ 3, 2] 2.06730E-01
[ 3, 3] 3.23219E-01
[ 3, 4] 3.08065E-01
[ 3, 5] 1.74949E-01
[ 3, 6] 8.99855E-02
[ 3, 7] 6.25087E-02
50000 7
[ 4, 1] 2.11506E-02
[ 4, 2] 8.66147E-02
[ 4, 3] 1.54937E-01
[ 4, 4] 1.91120E-01
[ 4, 5] 1.64706E-01
[ 4, 6] 1.30056E-01
[ 4, 7] 1.18124E-01
35000 7
[ 5, 1] 1.25065E-02
[ 5, 2] 5.27674E-02
[ 5, 3] 1.09205E-01
[ 5, 4] 1.63627E-01
[ 5, 5] 1.69904E-01
[ 5, 6] 1.49838E-01
[ 5, 7] 1.42506E-01
25000 7
[ 6, 1] 7.87436E-03
[ 6, 2] 3.42143E-02
[ 6, 3] 7.88815E-02
[ 6, 4] 1.31906E-01
[ 6, 5] 1.48247E-01
[ 6, 6] 1.36147E-01
[ 6, 7] 1.31627E-01
15000 7
[ 7, 1] 3.29592E-03
[ 7, 2] 1.54733E-02
[ 7, 3] 4.49785E-02
[ 7, 4] 8.93790E-02
[ 7, 5] 1.10891E-01
[ 7, 6] 1.06393E-01
[ 7, 7] 1.04560E-01De façon analogue, pour les observations de type
INTQTY,
la valeur
observée co est liée à l'état
modèle interpolé au point d'observation xm,
sur les niveaux du profile intermédiaire, par
la relation co = ca + A (xm - xa)
où xa est un profil à priori et A est une
matrice ligne (une par profil) de lissage et intégration dite averaging kernel. Puisque pour
l'assimilation nous sommes intéressés à la
différence entre valeur observée et valeur
estimée, il est plus économique de stocker dans le
fichier HDAT la quantité co - ca
+ Axa de façon
à la comparer directement
à la quantité Axm
produite par l'opérateur d'observation. La matrice A pour chaque profil est
stockée sous forme de matrice ligne dans
le fichier ASCII dont le nom commence par HAVK qui
contient les averaging kernels. La seule différence
réside dans le fait que, la matrice
étant réduite à un vecteur ligne, on aura un seul
niveau
pour lequel à la place du nombre d'espèces
observées on indique le nombre des niveaux du profil c'est
à dire le nombre
d'éléments du vecteur ligne A. A la place du nom de
l'espèce on indique les deux
indices de l'élément dans la matrice.La matrice associée au premier profil d'un fichier
HDAT relatif à une quantité
intégrée MOPITT avec a priori sur 7
niveaux verticaux est
stockée dans le fichier HAVK de la façon
suivante :
6214 1
MOPITT CO INTQTY=7
200307010012 MOPITT 00000 -36.49 -18.73 1
0 7
[ 1, 1] 8.32152E+01
[ 1, 2] 3.31613E+01
[ 1, 3] 5.02991E+01
[ 1, 4] 4.45117E+01
[ 1, 5] 2.08479E+01
[ 1, 6] 7.13190E+01
[ 1, 7] 2.74929E+01Installons le code!
On peut enfin attaquer les sources du modèle et de la chaîne d'assimilation.Après avoir extrait le contenu de
ADOMOCA_v_v_v.tgz
et de MOCAGE_CONSTANTS.tgz vous trouverez l'arborescence
suivante :README
ASSIMILATION/
MOCAGE_PALM/
MOCAGE_COMPILE_ASSIMILATION/
MOCAGE_JOBS/
MOCAGE_TOOLS/
Elle doit être complétée par les sources et les procédures de compilation du modèle direct (distribuées par le CNRM) :
MOCAGE/
MOCAGE_COMPILE_DIRECT/Et par les fichiers de constantes qui sont distribués dans l'archive
MOCAGE_CONSTANTS.tgz et qui contient le
répertoire :MOCAGE_CONSTANTS/Dans le détail, examinons le contenu de ces répertoires :
MOCAGE/- Le sous-répertoire
src_svcontient les fichiers sources du modèle direct communs aux quatre schémas. Grâce à l'utilisation du préprocesseur CPP, ces fichiers vont inclure des parties spécifiques à chaque schéma. Les sous-répertoiresCARIOLLE,REPROBUS,RELACS,RACMOBUScontiennent les parties spécifiques pour le schéma correspondant à leur nom MOCAGE_COMPILE_DIRECT/- Les sous-répertoires
src_CARIOLLE,src_REPROBUS,src_RELACS,src_RACMOBUScontiennent un include file (paradi_*) avec les paramètres de dimensionnement spécifiques pour la configuration choisie et leMakefilepour la compilation. Un script shell crée les liens symboliques vers les fichiers sources et les include files nécessaires contenus dans les précédents répertoires. MOCAGE_JOBS/- Contient le script de lancement
jobMCGelfautoexplicatif, à lancer en interactif sur PC ou sur Opteron et à soumettre à travers le filtremtoolsur le NEC. La terminaisonelfindique que l'auteur de cet outil très complet et pratique est Eric Le Flochmoën.
MOCAGE_TOOLS/- Contient les scripts auxiliaires appelés par le job de lancement.
MOCAGE_CONSTANTS/- Le sous-répertoire
chmcontient les paramètres des schémas linéaires sur la grille 2° x 2°, sur la grille 0,5° x 0,5° et sur la grille de Gauss et les paramètres pour les processus de surface pour les schémas RELACS et RACMOBUS. Le sous-répertoireclimcontient la fermeture climatologique au bord supérieur pour la configuration à 47 niveaux. Le sous-répertoiregausscontient les données de définition de la grille de Gauss T42.
Les répertoires suivants contiennent les fichiers pour la chaîne d'assimilation palmée.
ASSIMILATION/- Le répertoire
ASSIMILATIONcontient les fichiers sources des unités PALM propres à l'assimilation et indépendantes du choix du modèle. Le sous-répertoirePALM_IDCARDScontient les identity cards (au sens PALM) des unités. MOCAGE_PALM/- Le répertoire
MOCAGE_PALMcontient les include files (.h) qui permettent de transformer des routines MOCAGE en unités PALM. En plus on y trouve les fichiers (.ppl& al.) qui décrivent l'application palmée. Le sous-répertoirePALM_IDCARDScontient les identity cards (au sens PALM) des unités. MOCAGE_COMPILE_ASSIMILATION/- Les sous-répertoires
src_3DFGAT_CARIOLLE,src_3DFGAT_REPROBUS,src_3DFGAT_RELACS,src_3DFGAT_RACMOBUS,src_3DFGAT_NETCDFcontiennent un include file (paradi_*) avec les paramètres de dimensionnement spécifiques pour la configuration choisie et leMakefilepour la compilation. Un script shell crée les liens symboliques vers les fichiers sources et les include files nécessaires contenus dans les précédents répertoires. En plus ils contiennent deux versions (en correspondance des deux résolutions verticales) d'un fichier de paramètres de dimensionnement pour redéfinir les constantes PrePALM correspondantes. Le sous-répertoiresrc_TESTGRAD_CARIOLLEcontient ces mêmes fichiers plus les fichiers qui décrivent l'application palmée pour le test de validité du gradient codée à partir de l'application au modèle linéaire.
Le modèle direct
Compilation
On va commencer par apprendre à compiler le modèle direct.Nous pouvons distinguer de nombreuses versions en fonction du choix du schéma chimique et de la résolution verticale. Les différents exécutables sont obtenus à partir d'un jeu commun de fichiers sources par l'utilisation du préprocesseur CPP.
N.B. dans le cas de changement de clés CPP, se souvenir de réinitialiser la compilation en effaçant tous les fichiers objets (
make clean)Une clé CPP définit le schéma : il sera donc possible d'activer l'une de ces options
-DCARIOLLE
-DREPROBUS
-DRELACS
-DRACMOBUSCARIOLLE,
d'autres clés CPP doivent être utilisées pour
indiquer quelles espèces modéliser : il sera donc
possible d'activer une quelqconque combinaison de ces options (N.B.
au moins une clé doit être activée) -DLINO_3
-DLINCO
-DLINHNO_3
-DTFROID
Une paire de clés permet de choisir la
résolution
verticale :-DMIDATM :
l'activation de cette clé impose la prise en compte de la
moyenne atmosphère et impose le choix entre l'une des deux
clés suivantes-DNIV_60 : l'activation de
cette clé impose la discrétsation verticale à 60
niveaux-DNIV_91 :
l'activation de
cette clé impose la discrétisation verticale à 91
niveaux (pour le moment non utilisée dans le contexte ADOMOCA)
-DGAUSS : l'activation de
cette clé sélectionne la grille de Gauss globale en
troncature triangulaire T42-DPALM_DIAG est activée, les
diagnostics seront écrits dans des fichiers HPALM*
et, pour chaque observation, contiendront la valeur simulée par
le modèle au même point, la valeur de l'observation et
leur écart. Si cette clé n'est pas activée, les
diagnostics seront écrits dans des fichiers HDIAG*
et ne contiendront que les valeurs du modèle en correspondance
des observations. Le format HPALM* est
particulièrement pratique si l'intégration du
modèle sert de simulation de contrôle sur une
période
d'assimilation.Les clés
-DSBL et -DMASSy concernent
plus particulièrement les schémas
tropo-stratosphériques. La première active le traitement
simplifié de la couche limite et la deuxième un rappel
pour la conservations de la masse en cas de run long.Pour chaque schéma chimique la compilation aura lieu dans le répertoire correspondant
MOCAGE_COMPILE_DIRECT/src_SCHEMA.
Ainsi pour le schéma linéaire, quelle que soit la
résolution verticale on se positionnera dans MOCAGE_COMPILE_DIRECT/src_CARIOLLE.La première fois il faut exécuter le script
inst_links qui
crée dans le répertoire les liens vers les fichiers
sources et les include files de src_sv
et CARIOLLE (ou REPROBUS ou RACMOBUS
ou RELACS selon le schéma).La compilation se fait par
Makefile, avec, bien sûr
des commandes différentes en fonction de la plate-forme.La structure générale du
Makefile est
commune pour toutes les plates-formes et, normalement il n'est pas
nécessaire de la changer. Il inclût un fichier Make.commands
qui contient les commandes et les paths spécifiques pour chaque
machine. Vous trouverez déjà les Make.commands pour un PC linux (avec compilateur F90 du Portland Group ou avec le g95 gratuit), pour le Cray XD1 du CERFACS et pour le NEC de Météo-France. Ils ont une extension supplémentaire pour les distinguer (e.g.
Make.commands.pclinux)
: avant de l'utiliser il faudra renommer Make.commands le
fichier choisi.Voyons en détail le contenu de l'un de ces fichiers : le
Make.commands.pclinux
pour le schéma CARIOLLE sur plate-forme Linux avec compilateurs
PGI#
# MOCAGE compilation suite
v.1.0.0
# Makefile include file
Make.commands
# for the CARIOLLE linear
chemical scheme
# running on a Linux PC under
PGI
#
# Scheme selection and specific
dependencies
#
SCHEME=CARIOLLE
SCHEME_DEPS=cariop_mod.F
#
# Machine dependent inlining
#
TCVQSAT_OBJ=tcvqsat.o
TCVQSAT_INLINE=
#
# CPP keys
#
CPPFLAGS=-DCARIOLLE -DLINO_3 -DPALM_DIAG -DMIDATM -DNIV_60
#
# Compiler and linker commands
#
FC=pgf90
F90=$(FC)
LD=$(FC)
CC=
#
# Compilation flags as used in
the common Makefile
# (all of them should be
present even if empty or redefined)
#
# Generic fortran flags
FFLAGS=-r8 -pc 64 -byteswapio -O3 -Mdalign -Mextend
#
# Specific include paths
NCDFINC=
#
# Specific flags for NEC high
vector optimisation
FFLAGSX=$(FFLAGS)
#
# Specific flags for NEC low
vector optimisation
FFLAGS0=$(FFLAGS)
#
# Specific flags for safe (e.g.
IEEE) compilation
FSAFEFLAGS=$(FFLAGS)
#
# Speficic fine optimisation
tuning flags
F_EXTRAFLAGS=
#
# OpenMP activitation flag at
compile time
F_OMPFLAGS=-mp
C_OMPFLAGS=
#
# OpenMP activation flag at
link time
LD_OMPFLAGS=$(F_OMPFLAGS)
#
# Generic linker flags
LDFLAGS=$(FFLAGS) $(LD_OMPFLAGS) -L/usr/local/pgi/linux86/6.2/lib
-L/usr/local/pgi/linux86/6.2/lib -L/home/andrea/USERS/andrea/XRD
-L/home/andrea/USERS/andrea/GRIBEX/gribex_000263
#
# Linked libraries
LIBS=-lxrd_jfe -lgribexR64 -lnetcdf -lblas
Une fois le bon fichier
Make.commands mis en place, il ne
reste plus, si l'on est sur une machine où l'on compile en
interactif, qu'à tapermakejobMCGelf que l'on trouve dans le
répertoire MOCAGE_JOBS.
Aux alentours de la ligne 346 vous trouvez la cléCOMPIL=1
La valeur 1 active la compilation avant exécution, la
valeur 0 la désactive.Sur le NEC de Météo-France, la compilation doit nécessairement s'effectuer en batch.
On activera donc la clé
COMPIL du script de
lancement jobMCGelf que vous soumettrez, dans ce cas,
avec la commande
jobfilter.pl
jobMCGelfSi vous devez rajouter une nouvelle plate-forme, il vous faut créer le fichier
Make.commands
correspondant et de l'associer à un mot clé de votre
choix désignant votre environnement de compilation dans le
script compile_mocoge.sh du répertoire MOCAGE_TOOLS.
Ce mot clé devra apparaitre dans le script jobMCGelf du
répertoire MOCAGE_JOBS aux alentours de la ligne
405 ou l'on positionne la variable STATION=mot-clé.Il se pourrait que certains fichiers demandent des options de compilation différentes par rapport aux autres. C'est pour cette raison que les variable
FxxxFLAGSxxx ont
été introduites. Il vous faudra alors éditer le
fichier Makefile pour remplacer dans les lignes des
fichiers en question la variable FFLAGS générique
par la variable FxxxFLAGSxxx correspondante.Par exemple, pour le fichier
mtslbacktra.F90, qui doit
nécessairement être en arithmétique standard (ce
qui demande l'option -Kieee sur Cray), la ligne
correspondante estmtslbacktra.o : mtslbacktra.F cstes.h interp.h posi.h formt.h
forft.h form.h forc.h grids.h mocf.h expe.h paradi.h
$(FC) $(FSAFEFLAGS) $(CPPFLAGS) -c
mtslbacktra.F
Exécution
L'exécution de MOCAGE est pilotée par le scriptjobMCGelf
(dans le répertoire MOCAGE_JOBS) lequel s'appuie
sur une série de script rangés dans le répertoire MOCAGE_TOOLS.
Il gère les opérations de compilation, génération de la namelist, rapatriement des fichiers d'entrée dans le répertoire de travail, exécution, archivage des résultats sur différent plate-formes, y compris l'environnement "frontale+nœuds de calcul" NEC de Météo-France. Sur cette dernière machine toutes les phases s'exécutent en batch mais elles sont reparties sur les différents sous-systèmes par des directives
mtool
(cf. la documentation
utilisateur de la machine tori pour plus de
détail) ; sur les plates-formes interactives le même
script est lancé en ligne de commande.L'utilisateur doit renseigner le script sur les options de configuration, d'exécution (plus de détail dans quelques lignes), et sur les chemins d'accès aux données et à l'espace d'archivage.
Voilà la section "utilisateur" du script
jobMCGelf. Les
clés sont expliquées dans les commentaires du script
après chaque section. Ces commentaires sont
assez parlant ; si, toutefois vous avez d'autres questions,
n'hésitez pas à contacter lefe@aero.obs-mip.fr ou andrea@cerfacs.fr.#!/bin/ksh
#
#PBS -N ADO_3DFGAT
#
#MTOOL set VECTOR=torisx
#MTOOL set SCALAR=toritx
#
#MTOOL profile target=torisx
#PBS -N ADO_RUN
#PBS -S /bin/ksh
#PBS -T mpisx
#PBS -q vector
#PBS -j o
#PBS -b 1
#PBS -l cpunum_job=4
#PBS -l memsz_job=5000mb
#PBS -l cputim_job=06:00:00
#PBS -l elapstim_req=01:30:00
#MTOOL end
#
#MTOOL profile target=toritx
#PBS -N ADO_FT
#PBS -S /bin/ksh
#PBS -q ft
#PBS -j o
#PBS -l memsz_job=512mb
#PBS -l elapstim_req=00:30:00
#MTOOL end
#
#MTOOL profile target=toritx_compil
#PBS -N ADO_CMP
#PBS -S /bin/ksh
#PBS -q compile
#PBS -j o
#PBS -l elapstim_req=00:30:00
#MTOOL end
#
#MTOOL autolog
#MTOOL set logtarget=torisx
#MTOOL autoclean
#
#WAIT_QUEUE=$FTDIR
#MTOOL export WAIT_QUEUE=$MTOOL_STEP_WORKSPACE
#
#
################################################################################
#
# --- User parameters to use MOCAGE-PALM ---
#
# - Preparation, Compilation, Run, Archivage -
#
# Date : april 2007
# Autors : E. Le Flochmoen (LA), V.H. Peuch (CNRM), B.
Josse (CNRM)
#
# Date :
# Modification :
#
################################################################################
#
#
#============================================#
# 0. Parameters
for MOCAGE #
#============================================#
#
####################################################
# __ __ _____
_____ _____ _____ _____
# | \/ | / \
/ \ / _ \ / ___\ | ___|
# | | | | | |
<--< | _ | | \_ \ | ___|
# |_|\/|_| \_____/ \_____/ \_/ \_/ \_____/ |_____|
#
# MODELE DE CHIMIE ATMOSPHERIQUE A GRANDE ECHELLE
#
# VERSION 1.0
#
# Meteo-France
# Centre National de Recherches Meteorologiques
#
# - Preparation, Compilation, Run, Archivage -
#
# Original : V.-H. Peuch, GMGEC/ERAM, 08/1999
#
####################################################
#
#========================================#
# 1.
Pre-configuration #
#========================================#
#
MODE=BEST
MOCFG=DFLT
NSTART=1
INIT=CLIM
NIV=60
#---------------------------------------------------------------------
# MODE : type of forcings
#
if MODE=FCST, use forecasts (max. 72h)
#
if MODE=BEST, use best available forcings
#
(analyses and shot-term forecasts)
#
# MOCFG : MOCAGE configuration
#
if MOCFG=DFLT : GLOB22,EURO11,FRA025,RSE008 (DEFAULT)
#
if MOCFG=ACDT : GLOB22,EURAT5,FRA025,RSE008 (ACCIDENT)
#
if MOCFG=CITY : GLOB44,EMEP05,CITY01 (CITY-DELTA ; not yet!)
#
if MOCFG=T42 : GLOT42 (CLIMAT Gauss T42)
#
# NSTART : if NSTART =0, start a new simulation
#
if NSTART<>0, resume a previous run
#
# INIT : initialisation procedure
#
if INIT=CLIM, use 2D lat-lon
#
if INIT=ELI, use [Lary et al., 95] (not yet)
#
if INIT=TELI,use [Peuch et al., 99] (not yet)
#
# NIV : Number of levels in the forcing files (47 or 60)
#---------------------------------------------------------------------
#
#=========================================#
# 2. Namelist
parameters #
#=========================================#
#
DATEIN=2003070100
DATEOUT=2003070200
NDAYSPLIT=1
NHCY=6
NOUT=24
NDOM=1
CHEMSCHEME=CARIOLLE
SOLID=.TRUE.
if [ $CHEMSCHEME = CARIOLLE ] ; then
SURF=.FALSE.
PRMTRSP=NO
CLOUD=.TRUE.
fi
if [ $CHEMSCHEME = REPROBUS ] ; then
SURF=.FALSE.
PRMTRSP=NO
CLOUD=.TRUE.
fi
if [ $CHEMSCHEME = RELACS ] ; then
SURF=.TRUE.
PRMTRSP=KFB
CLOUD=.TRUE.
fi
if [ $CHEMSCHEME = RACMOBUS ] ; then
SURF=.TRUE.
PRMTRSP=KFB
CLOUD=.TRUE.
fi
OBS=.FALSE.
SRC=.FALSE.
TRANSINV=.FALSE.
NCDF=3
INICDF=1
CTOTCOL=1
CLEVELS=0
LEVELS="1:47"
CSHORT=0
SPECIES="CO O_x O_3 NO_2 N_2O"
#---------------------------------------------------------------------
# DATEIN : YYYYMMDDHH (begin)
#
# DATEOUT : YYYYMMDDHH (end)
#
# NDAYSPLIT : Number of days per jobs : used to split long jobs
#
# NHCY : frequency (h) for the
dynamical
#
forcing (ARPEGE : NHCY=6 or NHCY=3
#
# NOUT : frequency (h) for the
output of
#
history files
#
# NDOM : if NDOM=3
(Global+Contin.+Regional)
#
if NDOM=2 (Global+Continental)
#
if NDOM=1 (Global only)
#
# CHEMSCHEME : - CARIOLLE (Linearized Ozone strato chemistry)
#
- REPROBUS (Strato. chemistry only from [Lefevre et al.])
#
- RACMOBUS (REPROBUS+RACM from [Stockwell et al.])
#
- RELACS (Strato. + TROPO "light")
#
# SOLID : if .TRUE., take stratospheric
#
heterogeneous chemistry into account
#
# SURF : if .TRUE., compute surface
forcings (emissions
#
and/or deposition and/or evaporation). If
#
SURF=.FALSE., parameterized transport is not
#
taken into account
#
# PRMTRSP : parameterized transport
#
if PRMTRSP=TDK, use Tiedtke+Louis
#
if PRMTRSP=KFB, use Kain-Fritsch-Bechtold+Louis
#
if PRMTRSP=ARP, use ARPEGE schemes (not yet)
#
if PRMTRSP=NO , no diff./convec.
#
# CLOUD : if .TRUE., compute cloudiness
and apply
#
correction to photolysis rates, following [Chang, 1987]
#
# OBS : if .TRUE., use daily
HOBS* observational files and
#
ouput daily HDIAG* model equivalent
#
diagnostics files
#
# SRC : if .TRUE., take point
sources into account
#
(see detailed namelist below 0.6)
#
# TRANSINV : if .TRUE., compute in reverse "back-tracking"
mode
#
if .FALSE., compute in normal "forward" mode
#
# NCDF : frequency (h) for the
output of
#
NetCDF files
#
NCDF < 0 => NO NetCDF output
#
# INICDF : INICDF <> 0 => NetCDF
output of the restart
#
INICDF = 0 => No NetCDF output of the restart
#
# CTOTCOL : if CTOTCOL=1 and NCDF > 0 output total columns (2D) in
Netcdf
#
# CLEVELS : if CLEVELS=1 and NCDF > 0 only a subset of levels
# will be
output in NetCDF files
#
# LEVELS : list of output levels in NetCDF files (CLEVELS=1)
# Format : single blocks are separated by ;
#
blocks are l1[:l2[:l3]]
# for
levels from l1 to l2 with step l3
# if
l3 is not present step is 1
# if
l2 is not present the block corresponds to single level l1
#
exemple LEVELS="1:3;5:31:2;35;40;47"
# NB : if CLEVELS = 0 these indexes are neglected
# and the whole
vertical domain is output
#
# CSHORT : If NCDF > 0 :
#
if CSHORT=1 the selected species will be output
#
if CSHORT=0 only O_X (47NIV) or O_3 (60NIV)will be output
#
edit the CPP_ESP definition in src_sv/outnetcd.F to
#
change the default species
#
# SPECIES : Short list for post-treatment (if CSHORT=1) :
# names of
species
#
TYPE : character
# SYNTAX :
within double quotes and separated
#
by blank space(s)
# NB : names must match the species actually
defined
# within
$CHEMSCHEME
#---------------------------------------------------------------------
#
#======================================#
# 3. 1D model
(C1D=1) #
#======================================#
#
C1D=0
LON="10. 30. -1.0"
LAT="45. 1. -40.06"
#---------------------------------------------------------------------
# C1D : if C1D=1, use 1D-version
#
# LON : longitudes of columns
# TYPE : integer or real
# UNIT : degree
# RANGE : -180 to +180
# SYNTAX : within double quotes and
separated
#
by blank space(s)
#
# LAT : latitudes of columns
# TYPE : integer or real
# UNIT : degree
# RANGE : -90 to +90
# SYNTAX : within double quotes and
separated
#
by blank space(s)
#
# NB : (1) the number of longitudes and latitudes must
# be the same ;
longitudes and latitudes of
# columns must be
given in the same order ;
#
# (2) make sure $NAMEMAIN corresponds to
the 1D
# version of
MOCAGE.
#---------------------------------------------------------------------
#
#=================================================#
# 4. Point
sources ($SRC=.TRUE.) #
#=================================================#
#
PNAM="NAM> CESIUM CESIUM"
PLON="LON> -1.55 -1.55"
PLAT="LAT> 47.22 47.22"
PBOT="BOT> 0. -1000."
PTOP="TOP> -1000. -1300."
PQTY="QTY> 0.9E15 0.1E15"
PUNI="UNI> BQPERH BQPERH"
PDTB="DTB> 2002020512 2002020512"
PDTE="DTE> 2002020518 2002020518"
#---------------------------------------------------------------------
# SYNTAX FOR ALL ITEMS : list within double quotes
#
ex : "XXX> ITEM1 ITEM2 ITEM3"
#
# PNAM : Pollutants names (CHARACTER*15)
#
# PLON : Longitudes of the release (INTEGER or REAL ;
# degrees ; -180 to +180)
#
# PLAT : Latitudes of the release (INTEGER or REAL ;
# degrees ; -90 to +90)
#
# PBOT : Lower boundary of the release (REAL ; >0 if in Pa
# and <0 if in meters
above sea level)
#
# PTOP : Upper boundary of the release (REAL ; >0 if in Pa
# and <0 if in meters
above sea level)
#
# PQTY : Quantities released
#
# PUNI : Unit for released quantities (currently 'BQPERH')
#
# PDTB : Releases begin date AAAAMMJJHH
#
# PDTE : Releses end date AAAAMMJJHH
#---------------------------------------------------------------------
#
#==================================================#
# 5. Parameters
for assimilation #
#==================================================#
#
CASSIM=0
CNETCDF=0
TYPEOBS=MOPITT
ASSIMILATED_SPECIES="CO"
#----------------------------------------------------------------
# CASSIM : - if CASSIM=0 : MOCAGE is launched without
assimilation
# - if
CASSIM=1 : MOCAGE is launched taking account the assimilation with
PALM
#
# CNETCDF : - if CNETCDF=0 : the assimilation suite uses the direct
model
# - if
CNETCDF=1 : the assimilation suite uses a NetCDF loader instead
#
# TYPEOBS : observations used to do the assimilation
#
# ASSIMILATED_SPECIES : names of species which we assimilate with some
observations
#
TYPE : character
# SYNTAX :
within double quotes and separated
#
by blank space(s)
# NB : names must match the species actually
defined
# within $MODEL
(see file "list_species")
#----------------------------------------------------------------
#
#==============================#
# 6.
Controls #
#==============================#
#
CRUN=1
COLD=0
COMPIL=1
EXSAVE=0
MPTOOL=0
TOTAL=0
CFORC=0
CFORCS=0
CSURF=0
CSURFS=0
CPTGS=0
#---------------------------------------------------------------------
# CRUN : if CRUN=1, run a MOCAGE simulation else
# retrieve HM
files from $PATH_HM
#
# COLD : if COLD=1, retrieve (see section 0.4) history
files,
# forcings and /
or binaries from previous experiments
#
# COMPIL : if COMPIL=1 (and CRUN=1), compile MOCAGE
# sourcefile else
retrieve executable from $DIRSOURCES
#
# EXSAVE : if EXSAVE=1 (and COMPIL=1), write
# executable in
directory $PATHX
#
# MPTOOL : if MPTOOL=1 (and COMPIL=1), do
# performance
analysis
#
# TOTAL : if TOTAL=1, compile source with debugging
# option (-g3)
and run totalview ; make sure your
# $DISPLAY is set
properly
#
# CFORC : if CFORC=1, compute forcing files
# else, if
CRUN=1, retrieve them from $PATH_FM
#
# CFORCS : if CFORCS=1 (and CFORC=1), save
# forcing files
on $PATH_FM
#
# CSURF : if CSURF=1 (and SURF=.TRUE.), compute surface exchange
# files else, if
CRUN=1, retrieve them from $PATHS
#
# CSURFS : if CSURFS=1 (and CSURF=1), save surface
# exchange files
on $PATHS
#
# CPTGS : - if CPTGS=1, send history files onto
#
eram2:/mocage/databin for post-treatment
#
with GSHARP (binary files)
# - if CPTGS=2,
send surface files onto
#
eram2:/mocage/databin for post-treatment
#
with GSHARP (binary files)
# - if CPTGS=3,
send history as well as surface
#
files onto eram2:/mocage/databin for post-treatment
#
with GSHARP (binary files)
# NB : if CSHORT=1 (and CPTGS=1, 2 or 3), only
transfer
# a limited
number of chemical species for
# post-treatment
#---------------------------------------------------------------------
#
#===================================#
# 7.
Environnement #
#===================================#
#
##### 3.1 Stations and Paths ########################
#
STATION=tori
STATION_INPUT=DELAGE
STATION_OUTPUT=DELAGE
#
if [ $STATION = tori ] ; then
MOCAGE_ROOT=$HOME/MAQUETTE/ADOMOCA_V4
MUTILS=/cnrm/gc/mrgm/mrgm003/MUTILS
fi
if [ $STATION = aerosv1 ] ; then
MOCAGE_ROOT=$HOME/ASSIMILATION/DEV_ADOMOCA
fi
if [ $STATION = CERFACS ] ; then
MOCAGE_ROOT=/home/andrea/USERS/andrea/WORK/ADOMOCA_V4
fi
#
PATH_JOBS=$MOCAGE_ROOT/MOCAGE_JOBS
PATH_TOOLS=$MOCAGE_ROOT/MOCAGE_TOOLS
#
if [ $STATION_INPUT = DELAGE ] ; then
PATH_HM=/cnrm1/mrgm/mrgm205/ADOMOCA_V4/DATA/"$NIV"NIV/HM/"$CHEMSCHEME"
PATH_FM=/cnrm1/mrgs/mrgs509/MOCAGEFM/ECOPER
PATH_SM=/cnrm1/mrgm/mrgm205/ADOMOCA_V4/DATA/"$NIV"NIV/SM
PATH_OBS=/cnrm1/mrgm/mrgm203/MOCAGEOB/MIPAS_L60/200307L60
PATH_ASSIM=/cnrm1/mrgm/mrgm203/MOCAGEOB/MIPAS_L60/200307L60
PATH_JDATA=/home/m/mrgs/mrgs509/MOCAGECH
PATH_NC=$PATH_HM
fi
if [ $STATION_INPUT = aerosv1 ] ;
then
PATH_HM=/raid1/lefe/ASSIMILATION/DATA_MOCAGE/HM
PATH_FM=/raid1/lefe/ASSIMILATION/DATA_MOCAGE/FM/ARP
PATH_SM=/raid1/lefe/ASSIMILATION/DATA_MOCAGE/SM
PATH_OBS=/raid1/lefe/ASSIMILATION/DATA_OBSERVATIONS/${TYPEOBS}
PATH_ASSIM=/raid1/lefe/ASSIMILATION/DATA_ASSIMILATION/${TYPEOBS}/O3
PATH_JDATA=/raid1/lefe/ASSIMILATION/DATA_MOCAGE/CSTE
PATH_NC=$PATH_HM
fi
if [ $STATION_INPUT = CERFACS ] ;
then
PATH_HM=/home/andrea/SPACE/WORKSPACE/MOCAGE_V1/DATA/"$NIV"NIV/HM/"$CHEMSCHEME"
PATH_FM=/home/andrea/SPACE/WORKSPACE/MOCAGE_V1/DATA/"$NIV"NIV/FM
PATH_SM=/home/andrea/SPACE/WORKSPACE/MOCAGE_V1/DATA/"$NIV"NIV/SM
PATH_OBS=/home/andrea/SPACE/WORKSPACE/MOCAGE_V1/DATA/"$NIV"NIV/OBSERVATIONS
PATH_ASSIM=/home/andrea/SPACE/WORKSPACE/MOCAGE_V1/DATA/"$NIV"NIV/ASSIM
PATH_JDATA=/home/andrea/SPACE/WORKSPACE/MOCAGE_V1/DATA/PHOTO
PATH_NC=/home/andrea/SPACE/WORKSPACE/ADOMOCA_V4/RESULTS/"$NIV"NIV/DIRECT/"$CHEMSCHEME"_"$MOCFG"/`expr
$DATEIN | cut -c1-4`-`expr $DATEIN | cut -c5-6`fi
#
if [ $STATION_OUTPUT = DELAGE ] ;
then
if [ $CASSIM = 1 ] ; then
PATH_OUTPUT=ADOMOCA_V4/RESULTS/"$NIV"NIV/3DFGAT/"$CHEMSCHEME"_"$MOCFG"/"$TYPEOBS"-`expr
$DATEIN | cut -c1-4`-`expr $DATEIN | cut -c5-6`
else
PATH_OUTPUT=ADOMOCA_V4/RESULTS/"$NIV"NIV/DIRECT/"$CHEMSCHEME"_"$MOCFG"/`expr
$DATEIN | cut -c1-4`-`expr $DATEIN | cut -c5-6`
fi
fi
if [ $STATION_OUTPUT = aerosv1 ] ;
then
if [ $CASSIM = 1 ] ; then
PATH_OUTPUT=$MOCAGE_ROOT/OUTPUT/TESTS/${CHEMSCHEME}_${NIV}NIV_ASSIM
else
PATH_OUTPUT=$MOCAGE_ROOT/OUTPUT/TESTS/${CHEMSCHEME}_${NIV}NIV_DIRECT
fi
fi
if [ $STATION_OUTPUT = CERFACS ] ;
then
if [ $CASSIM != 0 ] ;
then
PATH_OUTPUT=/home/andrea/SPACE/WORKSPACE/ADOMOCA_V4/RESULTS/"$NIV"NIV/3DFGAT/"$CHEMSCHEME"_"$MOCFG"/"$TYPEOBS"-`expr
$DATEIN | cut -c1-4`-`expr $DATEIN | cut -c5-6`
else
PATH_OUTPUT=/home/andrea/SPACE/WORKSPACE/ADOMOCA_V4/RESULTS/"$NIV"NIV/DIRECT/"$CHEMSCHEME"_"$MOCFG"/`expr
$DATEIN | cut -c1-4`-`expr $DATEIN | cut -c5-6`
fi
fi
#
JOBID=EXEC
if [ $STATION = tori ] ; then
PATH_RUN=$TMP_LOC/$JOBID
PATH_WORK=$WORKDIR/$JOBID
fi
if [ $STATION = aerosv1 ] ; then
PATH_RUN=$MOCAGE_ROOT/$JOBID
PATH_WORK=$PATH_RUN
fi
if [ $STATION = CERFACS ] ; then
PATH_RUN=/home/andrea/SPACE/WORKSPACE/ADOMOCA_V4/$JOBID
PATH_WORK=$PATH_RUN
fi
#---------------------------------------------------------------------
# STATION : - if STATION=aerosv1 : PC linux opteron (LA)
# - if
STATION=CERFACS : PC linux (CERFACS)
# - if
STATION=tori : NEC (Meteo-France)
#
...
#
# STATION_INPUT : station where the HM, FM, SM and OBS files are stored
#
- at Meteo-France (STATION=tori) : STATION_INPUT=DELAGE
# STATION_OUTPUT : station where the results are stored
#
- at Meteo-France (STATION=tori) : STATION_OUTPUT=DELAGE
#
# MOCAGE_ROOT : directory of the MOCAGE distribution
#
(the one where the src_XXX directories can be found)
#
# PATH_JOBS : directory where the jobs are stored
# PATH_TOOLS : directory where some tools used in a pre-treatment of
#
MOCAGE are stored
#
# PATH_HM : directory where the history files are stored
# PATH_FM : directory where the forcing files are stored
# PATH_SM : directory where the surface exchange files are stored
#
# PATH_OBS : directory where the observation files are stored (HOBS*)
# PATH_ASSIM : directory where the files for assimilation are stored
(HDAT*,HCOV*,HAWK*)
# PATH_JDATA : directory where the photolyse files are stored (Jdata*)
#
# PATH_OUTPUT : directory where the results are stored
#
# JOBID : name used to identify the job
# PATH_RUN : directory where MOCAGE is runing
# PATH_WORK : directory where some using files are stored
#----------------------------------------------------------------
#
#===================================================================#
# 8. climatology
and previous experiments
(COLD=1) #
#===================================================================#
#
PATHC=DELAGE
CSAVEH=1
CSAVEF=0
CSAVES=0
CSAVEX=1
#---------------------------------------------------------------------
# PATHC : chemistry,J,clim2d (read)
# - if PATHC='DELAGE'
use ftput/ftget instead of cp
# - directory for
climatologies on tori : /cnrm1/mrgs/mrgs522/MOCAGECH/
#
# CSAVE* : if CSAVE*=1 (* = H, F, S or X), copy
#
the previous experiment data into the
#
directories for the current experiment
#
# NB : (1) if COLD<>1, nothing is done ;
#
# (2) not all the files of the previous
experiments
# are retrieved :
namelist and controls apply ;
#
# (3) if CSAVE*<>1, files are only
retrieved on a
# temporary
directory ; they are dismissed once
# jobMCG is over.
#---------------------------------------------------------------------
#
#===============================================#
# 9. Job
management parameters #
#===============================================#
#
JOBNAME=jobMCGelf
CSMAIL=0
USRMEL="andrea@cerfacs.fr"
#---------------------------------------------------------------------
# JOBNAME : name of the present file
#
# CSMAIL : if CSMAIL=1, send e-mail to $USRMEL
# upon completion
of the whole job
#
# USRMEL : user's e-mail
#---------------------------------------------------------------------
Si tout va bien il n'y plus rien à changer plus bas pour l'exécution monoprocesseur. Dans le cas parallèle OpenMP, le choix du nombre de "threads" se fait par positionnement d'une variable d'environnement dans le script
run_mocage.sh du
répertoire MOCAGE_TOOLS. La variable
d'environnement dépend du compilateur : sur le NEC de
Météo-France c'est OMP_NUM_THREADS, tandis
qu'avec les compilateurs PGI, c'est NCPUS. Dans le cas de
soumission en batch, il faut vérifier que le nombre de threads
soit cohérent avec le nombre de processeurs alloués.Pour le lancement sur PC ou en interactif sur Opteron, il suffira de lancer la commande
jobMCGelf.Sur NEC, après avoir adapté les options de soumission, on soumettra le job avec en utilisant le filtre de
mtool par
la commande jobfilter.pl jobMCGelf.Trois remarques sur cette procédure :
- La durée globale de la simulation est
déterminée par les dates
DATEINetDATEOUT. Toutefois, si la période de simulation est trop longue, il est impossible de la couvrir par une seule intégration (stockage des fichiers en entrée et en sortie, ressources batch, risque de plantages intermédiaires). Dans ce cas on fractionne la simulation en une série d'intégrations plus courtes. La variableNDAYSPLITindique le nombre de jours à couvrir avec chaque intégration. Par exemple, siDATEIN=20030701,DATEOUT=2003080100,NDAYSPLIT=8, la période de 31 jours est couverte par 3 intégrations de 8 jours et une de 7 pour compléter le mois.
- La variable
JOBID, sert à définir un espace de travail sur disque privé pour ce job. Ceci permet de lancer plusieurs jobs pour les mêmes dates sans interférence : il suffit d'indiquer desJOBIDdifférents. C'est par ce mécanisme que l'on gère facilement des simulations d'ensemble.
- Pour ajouter une plate.forme de travail (cf. le cas de la
compilation) il faut lui associer un mot clé. Les
procédures de transfert de fichiers et de lancement
correspondantes doivent être décrites dans les scripts du
répertoire
MOCAGE_TOOLSet la plate-forme doit être déclarée dans le scriptjobMCGelf. Pour trouver les endroits à modifier, il suffit de chercher le chaîneSTATIONdans tous les scripts du répertoireMOCAGE_TOOLS(grep -in STATION MOCAGE_TOOLS/*)
ATTENTION : une mesure de prudence, empêche à MOCAGE d'écraser des fichiers au format Arpège. Il est donc impératif qu'il n'y ait aucun autre fichier de type
HMGLOB22+date au-delà de la
condition initiale HMGLOB22+2003070100 dans le
répertoire
d'exécution.A la fin de l'exécution vous trouverez dans le répertoire de stockage des résultats les fichiers suivants :
exp_description.log####################################################### __ __ _____
_____ _____ _____ _____
## | \/ | / \
/ \ / _ \ / ___\ |
___| ## | | | | |
| <--< | _ | | \_ \ | ___| ## |_|\/|_| \_____/ \_____/ \_/ \_/ \_____/ |_____| ##
####################################################### END OF JOB : 2003070100 TO
2003070200
EXPERIMENT : DIRECT
RUN
Scheme RACMOBUS
60 levels RESULTS STORED on DELAGE:
MOCAGE_V1/RESULTS/60NIV/DIRECT/RACMOBUS
#######################################################
####| Version Maquette ADOMOCA | CNRM-CERFACS & al |### #
#######################################################Parameters: Forcing reading frequency (NHCY): 6Number of domains (NDOM): 1Heterogeneous chemistry (SOLID): .TRUE.Surfaces processes (SURF): .TRUE.Parametrized transport (PRMTRSP): KFBClouds (CLOUD): .TRUE.Obs file HOBS+20030701ContainingMIPAS OZONE PROFILE namelist+datein+dateoutparadi_SCHEMAnDOM+datein+dateoutHMGLOB22+2003070100.ncNCDF >
0 et INICDF = 1HMGLOB22+2003070106.nc
HMGLOB22+2003070112.nc
HMGLOB22+2003070118.nc
HMGLOB22+2003070200.ncNCDF. Dans ce cas NCDF
= 6HMGLOB22+2003070200NOUT et en tout cas à la date finale DATEOUT
(restart)HDIAGGLOB22+20030701+W00OBS
= .TRUE.). Il s'appellerait HDIAGGLOB22+20030701 si la clé CPP -DPALM_DIAG
n'était pas activée à la compilationJuste une petite remarque sur les fichiers NetCDF et Arpège : si pour la configuration CARIOLLE 47NIV un fichier restart Arpège "ne fait que" 24Mo, pour RACMOBUS 60 niveaux il arrive a 376Mo. Dans les sorties NetCDF vous choisissez quels champs écrire (cf. les entrées
CSHORT et SPECIES dans
jobMCGelf), vous pouvez choisir de limiter les sorties
à un sous-ensemble de niveaux (cf. les entrées CLEVELS
et LEVELS) et, enfin, parmi les champs dynamiques
on ne garde que la pression de surface (2D), les coefficients a et b de la coordonnées sigma
(1D, qui avec la pression de surface permettent de reconstruire la
pression 3D), la température et les trois composants
du vent. De plus, les champs sont
représentés en REAL(KIND=4). Par exemple,
pour les versions à 60 niveaux, en sortant tous les niveaux plus
la colonne totale (2D) pour 3 espèces on obtient un fichier de
22Mo.La chaîne d'assimilation
Le schéma PrePALM
L'application avec assimilation est implémentée avec le coupleur PALM (plus précisément avec la version PALM_RESEARCH). Le coupleur PALM est distribué gratuitement pour des applications recherche, après signature d'un accord fixant les termes d'utilisation. Pour obtenir le code il faut contacter le groupe PALM au CERFACS : palm@cerfacs.fr. Le logiciel est accompagné d'un guide de l'utilisateur en ligne qui comprend aussi un tutorial en 10 leçons.Périodiquement le CERFACS organise des formations aux nouveaux utilisateurs.
Pour cette raison nous ne détaillerons pas les aspects liés à l'utilisation de PALM mais nous vous invitons à consulter le guide de PALM (en particulier le chapitre Glossary and Quick Reference Guide) pour les doutes relatifs au coupleur.
N.B. pour les utilisateur du système NEC de Météo-France, il est possible d'exécuter l'interface graphique PrePALM directement sur la frontale tori, mais seulement dans un job en batch. Le script de lancement
jobPP (dans le répertoire MOCAGE_JOBS)
sert à lancer l'interface sur toriSans refaire la théorie du 3D-FGAT voyons quel est le principe de l'algorithme, de façon à comprendre, selon les lignes générales, le rôle des unités. Pour suivre cette partie il est conseillé de garder sous la main une fenêtre PrePALM ouverte sur le fichier
MOCAGE_PALM/MOCAGE.ppl.N.B. pour éviter des mauvaises surprises plus tard, songez à vérifier que vous avez au moins la version 2.2.5 de PrePALM (menu Help, commande About PrePALM), sinon, écrivez à palm@cerfacs.fr.
3D indique que nous cherchons une correction dans l'espace des
variables contrôlées, mais sans dépendance du
temps. Ceci signifie que nous cherchons une correction  x
x 

 xi qui peut être
appliquée à la trajectoire entière du
modèle. Dans d'autres termes, nous cherchons une "petite"
translation de la trajectoire qui la rapproche le "mieux"
possible aux observations.
xi qui peut être
appliquée à la trajectoire entière du
modèle. Dans d'autres termes, nous cherchons une "petite"
translation de la trajectoire qui la rapproche le "mieux"
possible aux observations.
FGAT est un acronyme de "First Guess at Appropriate Time". Ceci signifie que, même si la correction ne dépend pas du temps, les observations sont prises en compte avec leur temps exact et comparées avec l'ébauche ("first guess") xib au temps correspondant.
L'ébauche sur la fenêtre d'assimilation est le
résultat d'une intégration du modèle à
partir de la condition initiale x0b lue dans le
fichier de restart
xib= Mi,0(x0b)
C'est le rôle de l'unité INIT (lecture
du restart et initialisation des champs du modèle) suivie de
l'unité MODEL qui
fait avancer en temps le modèle.
Les données d'observation sont lues dans les fichiers HDAT,
HCOV, HAVK au
format ascii des observations MOCAGE. Un chargeur doit s'occuper de les
lire, de sélectionner celles qui tombent à
l'intérieur de la région spatio-temporelle
relative à la fenêtre d'assimilation et de remplir les
vecteurs d'observations yi0, les matrices de
variance/covariance
Ri-1
et celles des averaging kernels Ai. C'est
le rôle
de l'unité LECTOBS.
L' unité INIT_OBS
doit être exécutée au préalable pour
initialiser les paramètres et les tableaux des chargeurs.
Les observations ne correspondent pas
nécessairement à l'état du modèle.
Pour comparer les champs simulés et les observations nous avons
besoin d'un opérateur pour convertir l'état du
modèle dans une quantité observable qui lui correspond.
C'est l'opérateur d'observation
Hi associé à l'observation yi0.
Il peut comprendre une partie de calcul d'une quantité
dérivée (e.g. une espèce non pronostique, ou une
quantité intégrée), la colocalisation en temps
avec l'observation, la colocalisation en espace sur l'horizontale, la
colocalisation sur la verticale et un lissage (averaging kernel).
L'unité correspondante à Hi est
H.
Puisque les opérateurs H
agissent sur une grille de Gauss, les champs du modèle, si
nécessaire, doivent être interpolés sur une grille
de Gauss. C'est le rôle de unites model2gauss_ini
et model2gauss.
Avec cet opérateur on calcule l'écart di,
("misfit") entre champs du modèle et observations :
di = yi0 -
Hi(xi) = yi0
- Hi
(Mi,0(x0))
WRTOBS écrit
dans les
fichiers diagnostics ascii HDOM* et dans les fichiers
NetCDF HSTATS* le misfit.L'écart di calculé pour l'ébauche est archivé .
Nous avons dit que le but du 3DFGAT est de trouver une correction constante pour la
 x trajectoire du
modèle.
x trajectoire du
modèle.Nous voulons évaluer comment l'écart di change quand la correction
 x est
appliquée.
x est
appliquée.La nouvelle expression de l'écart est d'i = yi0 - Hi(xi +
 x).
x).Si nous linéarisons l'opérateur d'observation et nous indiquons l'opérateur linéarisé avec Hi, nous pouvons récrire l'expression précédente :
d'i = yi0 - Hixi - Hi
 x = di
- Hi
x = di
- Hi x
x Cette formulation nous permet de calculer l'écart di
relatif à l'ébauche une seule fois, de
l'archiver dans
le buffer de PALM (champs rda_dg) et ensuite de calculer
d'i à chaque itération de la minimisation en
appliquant l'opérateur linéarisé Hi
à l'incrément  x.
x.
L'unité de l'opérateur linéarisé est H_LT
est le calcul de d'i (par commodité on calcule -d'i)
est effectué par l'unité algébrique PALM algebra_3.
Nous construisons une fonction coût qui exprime sous forme
mathématique que nous cherchons un incrément  x qui réduise l'écart
entre
modèle et observations mais qui soit en même temps assez
petit pour que la nouvelle solution soit assez proche de
l'ébauche. Pour cette raison, la fonction coût est
composée de deux termes. Les deux termes sont
pondérés par les matrices de variance/covariance des
erreurs respectives. Puisque la taille de l'état du
modèle est trop grande pour construire la matrice B de
variance/covariance de l'erreur sur l'ébauche, le produit
matrice
vecteur est approximé par un opérateur linéaire de
type "diffusion" (cf. A.WEAVER;
Ph. COURTIER, Correlation
modelling on the sphere using a generalized diffusion equation, QJRMS,
July 2001 Part A, vol. 127, no. 575, pp. 1815-1846(32) ).
x qui réduise l'écart
entre
modèle et observations mais qui soit en même temps assez
petit pour que la nouvelle solution soit assez proche de
l'ébauche. Pour cette raison, la fonction coût est
composée de deux termes. Les deux termes sont
pondérés par les matrices de variance/covariance des
erreurs respectives. Puisque la taille de l'état du
modèle est trop grande pour construire la matrice B de
variance/covariance de l'erreur sur l'ébauche, le produit
matrice
vecteur est approximé par un opérateur linéaire de
type "diffusion" (cf. A.WEAVER;
Ph. COURTIER, Correlation
modelling on the sphere using a generalized diffusion equation, QJRMS,
July 2001 Part A, vol. 127, no. 575, pp. 1815-1846(32) ).
Les variances et covariances d'erreur peuvent être
approximées par proportionnalité au champs
d'ébauche, ou estimées par des statistiques d'ensemble et
lues dans des fichiers NetCDF. C'est l'unité INISTATS qui
construit les coefficient pour B
en fonction de la modalité de modélisation choisie.
La fonction coût et son gradient sont :
min ½ || x||B2
+ ½
x||B2
+ ½  ||di
- Hi
||di
- Hi x
||R2
x
||R2
min ½  xT
B-1
xT
B-1
 x +
½
x +
½  (di - Hi
(di - Hi x)T
Ri-1
(di - Hi
x)T
Ri-1
(di - Hi x)
x)
grad x J(
x J( x) = B-1
x) = B-1 x +
x +
 - (HiT
Ri-1
(di - Hi
- (HiT
Ri-1
(di - Hi x + di))
x + di))
Dans la pratique, le problème est reformulé avec un
changement de variable pour améliorer le conditionnement et
rendre le calcul plus efficace. Puisque l'ébauche domine le
problème, la Hessienne de la fonction coût peut
être
approximée par
B-1.
En admettant de savoir définir la racine carrée de
l'opérateur
B, nous pouvons introduire v t.q.
v = B-½ x
x
 x =
B½v
x =
B½v
Ce sera l'unité sqrt_B qui
effectuera le passage de v à  x.
Elle s'appuie sur une méthode spectrale pour la solution
de l'équation de diffusion qui donne les meilleurs
résultats sur une grille de Gauss. Les champs modèle
(pression, statistiques d'erreur) sont interpolés sur la grille
de Gauss par l'unité
x.
Elle s'appuie sur une méthode spectrale pour la solution
de l'équation de diffusion qui donne les meilleurs
résultats sur une grille de Gauss. Les champs modèle
(pression, statistiques d'erreur) sont interpolés sur la grille
de Gauss par l'unité p_sigma_to_gauss.
La fonction coût et son gradient par rapport à la nouvelle variable sont:
J = ½ vTv + ½
 (di - HiB½v)T
Ri-1
(di - HiB½v)
(di - HiB½v)T
Ri-1
(di - HiB½v)
gradv J(v) = v +
B½T
 - (HiT
Ri-1
(di - HiB½v))
- (HiT
Ri-1
(di - HiB½v))
Dans les faits, en partant du vecteur de contrôle v (mis
à
zéro initialement par l'unité INITv
et
fourni ensuite par le minimiseur algebra_7)
nous pouvons
facilement obtenir les termes de J relatifs à
l'ébauche
Jb = ½ vTv
(unité algebra_6)
grad Jb = v (l'une des deux
entrées de algebra_2)
Pour calculer les termes relatifs aux observations nous devons
calculer
 x =
B½v (unité
x =
B½v (unité sqrt_B)
ensuite
 yi =
HiB½v
(unité
yi =
HiB½v
(unité H_LT)
et la mise à jour de l'écart
- d'i =  yi -
di (unité
yi -
di (unité algebra_3)
Le "dual" de l'écart
d'i* =
Ri-1d'i
est calculé par l'unité INV_R.
Et enfin on complète la fonction coût avec la composante
Jo = ½  d'iT
d'i* (unité
d'iT
d'i* (unité algebra_4)
qui est rajoutée (J = Jb + Jo)
directement dans le buffer PALM.
Pour compléter le gradient il faut faire entrer en jeu les
adjoint
des opérateurs Hi et B½
dans la formule
grad Jo = B½T
 HiTd'i*
HiTd'i*
Ils correspondent aux unités Hstar et sqrtB_T
Le gradient
grad J = grad Jb + grad
Jo
est reconstitué par l'unité algebra_2
qui le passe au
minimiseur (algebra_7)
A la fin de la minimisation, la solution va est
transformée en incrément d'analyse  xa par l'unité
xa par l'unité sqrtB_fcst et
interpolé sur la grille modèle, si nécessaire, par
l'unité increment_to_model.
L'incrément est
sauvegardé dans un fichier NetCDF (DXGLOB22+date)
par l'unité plot_inc et
ensuite on itère sur la prochaine fenêtre d'assimilation
avec
une nouvelle intégration de MODEL, calcul
de l'écart etc...
Après la dernière fenêtre, le modèle pur
est
intégré jusqu'à la date DATEOUT de
la namelist pour la sauvegarde du restart analysé (unité MODEL_fcst).
Pour compléter la description de l'algorithme il y a un certain nombre de paramètres qui sont fixés sous forme de constantes PrePALM (Menu Constants, commande User constant editor). On les retrouve ensuite comme constantes PALM dans les fichiers palm_user_param.f90 et palm_user_param.h.
N.B. Ces constantes sont décrites avec plus de détail dans le document Constantes_PrePALM.html.
INTEGER, PARAMETER :: ip_lonm1 = 180 ! Number of
model grid longitudes
INTEGER, PARAMETER :: ip_latm1 = 90 ! Number of model
grid latitudes
INTEGER, PARAMETER :: ip_nivm1 = 60 ! Number of model
grid vertical levels INTEGER, PARAMETER :: ip_lonc1 = ip_lonm1 ! Number of
control grid longitudes
INTEGER, PARAMETER :: ip_latc1 = ip_latm1 ! Number of control
grid latitudes
INTEGER, PARAMETER :: ip_modelgrid_angles = 1 ! Model grid
angles unit : 1 for radians, 0 for degrees
INTEGER, PARAMETER :: ip_vert_coord = 1 ! Vert. coord scheme : 1
for hybrid sigma, 0 for pure pressure
INTEGER, PARAMETER :: ip_periodic_lon = 1 ! Periodic longitudes
: 1 for periodic, 0 for limited area
INTEGER, PARAMETER :: ip_lat_no_poles = 1 ! Latitude grid and
poles: 1 if poles not included (e.g. MOCAGE), 0 if poles are included
(e.g. LMDz)
INTEGER, PARAMETER :: ip_gauss_modelgrid = 0 ! Model grid type:
0 if regular,1 if Gauss (e.g. MOCAGE T42) INTEGER, PARAMETER
:: ip_ntra1 =
1 ! Number of transported
species
INTEGER, PARAMETER :: ip_nsls1 = 0 ! Number of short-lived
species INTEGER, PARAMETER :: ip_nassim = 1 ! Number of
assimilated
transported species INTEGER, PARAMETER :: ip_ninstmax = 1 ! Max
number of instruments for one species INTEGER, PARAMETER ::
ip_ninstmax_avk = 1 ! Max
number of instruments with avg. kernel (AVKERN) INTEGER, PARAMETER
:: ip_nobsmax = 1800 ! Maximum number of
observation each day
INTEGER, PARAMETER :: ip_nobstspmax = 180 ! Maximum number of
observation each slot
INTEGER, PARAMETER :: ip_nobsperinst_noavk_slot = 180 ! Maximum
nb of obs in a slot per instr. without av. kern.
INTEGER, PARAMETER :: ip_nobsperinst_avk_slot = 0
! Maximum nb of obs in a slot per instr. with av. kern. INTEGER, PARAMETER :: ip_nbnivpresmax = 18 ! Maximum
number of
pressure level
INTEGER, PARAMETER :: ip_nbniv_avk_in = 7 ! Maximum
number of levels before application of avg. kern.
INTEGER, PARAMETER :: ip_nbniv_avk_out = 7 !
Maximum number of levels after application of avg. kern.
INTEGER, PARAMETER :: ip_tc_as prof = 1 ! Tot col
treatment: 0 as scalar, 1 as shifted profiles
TOTCOL : elles peuvent être
assimilées comme des scalaires (H
contient une sommation) ou comme des profils équivalents obtenus
par modification du vecteur d'ébauche
INTEGER, PARAMETER :: ip_latobsmin = -90 !
Observations
south of
this latitude are discarded
INTEGER, PARAMETER :: ip_latobsmax = 90 ! Observations north
this
latitude are discarded
INTEGER, PARAMETER :: ip_lonobsmin = -180 !
Observations west
of this lon. are discarded. Use [-180, 180] period
INTEGER, PARAMETER :: ip_lonobsmax = 180 ! Observations east
this
lon. are discarded. Use [-180, 180] period
INTEGER, PARAMETER :: ip_presobsmin = 0 !
Observations over
this pressure (Pa) are discarded
INTEGER, PARAMETER :: ip_presobsmax = 150000 ! Observations
under this pressure (Pa) are discarded INTEGER, PARAMETER :: ip_enforced_obs_min = 0
! Min enforced observation value **in ppb** INTEGER, PARAMETER :: ip_tshold
= 200 !
Percentual misfit rejection threshold. 0 to take all INTEGER, PARAMETER :: ip_sigobs = 100 !
Percentual scale
factor of obs.
error covariances HCOV INTEGER, PARAMETER
:: ip_enforced_ana_min = 0 ! Min enforced analysis field
value **in ppb**
INTEGER, PARAMETER ::
ip_Bvar_rep = 1 ! Bkgr. err. var. representation: 1=computed from
model(by ip_sigbck), 2=1D constant per level(from file), 3=full 3D(from
file)
INTEGER, PARAMETER :: ip_sigbck
= 20 !
Percentual rms for background error
ip_Bvar_rep
= 1
INTEGER, PARAMETER ::
ip_Bhcor_rep = 1 ! Bkgr. err. hor. corr.
representation: 1=constant(by ip_flsh), 2=1D constant per
level(from file), 3=full 3D(from file)
INTEGER, PARAMETER
:: ip_flsh = 4 ! Hor.
bckgr. err. correlation length
scale (in degs.) (4) ip_Bhcor_rep
= 1
INTEGER, PARAMETER
:: ip_scrip = 1 ! Grid remapping: 1 for SCRIP, 0 for SPHEREPACK
INTEGER, PARAMETER :: ip_Bvcor_rep = 1 ! Bkgr. err. vert. corr.
representation: 1=constant(by ip_flsh), 2=1D vertical varying, equal
for all locations(from file), 3=full 3D(from file)
INTEGER, PARAMETER :: ip_flsz = 4 ! Vert.
bckgr. err. correlation
length
scale (in log(pressure)) (4) ip_Bvcor_rep = 1
INTEGER, PARAMETER ::
ip_vcor_1Dapprox = 1 ! Vert. coord appr. for vert.
correl. 1=1D av.profile (faster, ok for strato), 0=full 3D (better for
tropo) INTEGER, PARAMETER
:: ip_nbfenet = 8 ! Number
of
assimilation
windows INTEGER, PARAMETER :: ip_fenetre = 3*60*60 ! Size
(in sec.) of
each assimilation window INTEGER, PARAMETER :: ip_obs_slot = 1*60*60 ! Observ. time
slot i.e. frequency of model puts (secs.) INTEGER, PARAMETER :: ip_correctime =
ip_fenetre * 0/ 3 !
Time
where the correction is added INTEGER, PARAMETER :: ip_insincr = 0 ! if
ip_insincr = 1,
the
increment is added linearly over the assimilation windows INTEGER, PARAMETER :: ip_niter = 100 ! Max
number of iterations of the minimizer INTEGER, PARAMETER :: ip_omf = 1 ! ip_omf = 1
to output obs. minus forecast diags INTEGER, PARAMETER :: ip_oma = 1 ! ip_oma =
1 to output obs. minus analysis diags INTEGER, PARAMETER :: ip_omd = 1 ! ip_omd =
1 to output obs. minus dry run spanning the whole assim. exp. diags
HDOMF+,
HDOMA+, HDOMF+ et dans le fichier NetCDF HSTAT+.
Si la clé ip_omd est activée, le
modèle est intégré la
première fois de DATEIN à DATEIN +
ip_assimsize pour créer la
trajectoire de contrôle. INTEGER, PARAMETER
:: ip_nproc = 1 ! Number of processors
for the direct parallel model
Nombre de processeurs pour le modèle parallèle :
attention la clé CPP -DMPI doit être
activée à la compilation INTEGER, PARAMETER :: ip_nproc_B = 2 ! Number
of processors
for the direct parallel model
Nombre de processeurs pour le modèle parallèle :
attention la clé CPP -DMPI doit être
activée à la compilation INTEGER, PARAMETER ::
ip_restart_on_dsk = 0 ! if = 0 keep
old i.c. in palm, if =1 store it on disk
Méthode de sauvegarde des restart, par buffer PALM (plus rapide)
ou sur disques (plus économique en mémoire) INTEGER, PARAMETER :: ip_iprint = 0 ! Toggle
diagnostic output INTEGER, PARAMETER :: ip_loader_verbosity = 0
! Obs loader verbosity : 0 = silent, 1 = rejection info, 2 = complete
info INTEGER, PARAMETER :: ip_assimsize =
ip_nbfenet*ip_fenetre !
Size (in sec.) of global assmilation window ip_nbfenet*ip_fenetre) mais elle peut être plus longue si l'on
veut que la dernière intégration du modèle (la
partie forecast) aille au-delà de la période
d'assimilation INTEGER, PARAMETER :: ip_lono1 =
ip_lonc1 ! Number of observable grid longitudes
INTEGER, PARAMETER :: ip_lato1 = ip_latc1 ! Number of observable
grid latitudes INTEGER, PARAMETER :: ip_dimx = formule ! Vector size of
transported
species INTEGER, PARAMETER :: ip_dimobs = ! Dimension of
observation
space
INTEGER, PARAMETER :: ip_dimcovobs = ! Dimension of
covariance space for observations
INTEGER, PARAMETER :: ip_tracer_size = ! Storage size of a
tracer INTEGER. PARAMETER :: ip_H_size = ! Storage size of
csr H matrices
INTEGER, PARAMETER ::
ip_buff_size = formule
! Palm buffer size
Calcul automatique de la taille du buffer (la formule peut etre
retouchée en fonctions des machines)A partir de la version 2.2.3 de PrePALM il est possible de lire dans un fichier qui ait le même format de
palm_user_param.f90
un sous-ensemble des paramètres (Menu Constant,
commande Load constants from
file). PrePALM demandera une confirmation pour remplacer
les variables déjà définies et dont la valeur
change. Pour adapter les paramètres au nombre d'espèces
de chaque schéma, dans chaque répertoire src_3DFGAT_SCHEMA, des fichiers palm_schemenameXX_param.f90
contiennent les dimensionnements relatifs au schéma schemename
à XX niveaux. Ainsi, par exemple, palm_repro60_param.f90
contient les paramètres pour la configuration REPROBUS à
60 niveaux.A partir de la version 2.2.4 de PrePALM quand on change de configuration la taille mémoire du buffer interne PALM est réglée automatiquement. Toutefois la taille nécessaire peut changer en fonction de la stratégie d'allocation de chaque machine. Il est recommandé de vérifier à l'aide de l'analyseur de performances la réelle occupation mémoire et de modifier la formule du paramètre
ip_buff_size dans les
constantes PALM (Menu Constants, commande
User constants editor...,
ligne ip_buff_size).Vous êtes prêts pour la génération des fichiers PrePALM
Si vous changez seulement la taille du buffer ou quelques attributs des communications ou d'exécution, il vous suffit de régénérer le fichier
MOCAGE.pil.Si vous avez changé les constantes dont la case "Export" est cochée, il vous faudra régénérer les deux fichiers de paramètres
palm_user_param.f90
et palm_user_param.h.Si vous avez changé quelque chose dans le codage des branches vous devrez régénérer les fichiers de branche
Branch_one.f90, Branch_obs.f90Si vous avez changé de nom au fichier
.ppl (et
donc .pil) vous devrez régénérer le
fichier palm_init.f90.Enfin si vous avez ajouté ou remplacé des unités il vous faudra un nouveau fichier
palm_trigger.F90. N.B. PrePALM génère
palm_trigger.f90 (Petit f90!)
Le .F90 est obtenu à la main en remplaçant
les appels BLAS simple précision (e.g. SDOT, SAXPY,
...) par les correspondants en double précision (DDOT,
DAXPY, ...) Ceci est dû à une limite des
compilateurs lors de la promotion des réels : les variables
passent de 4 à 8 bytes, mais les appels aux fonctions restent
codés pour 4 bytes. Comparez le
palm_trigger.f90 et le palm_trigger.F90
qui sont distribués ; par exemple, ligne 442 du .f90
vs. la 444 du .F90 :palm_trigger.f90
CALL saxpy(N, alpha, X, incx, Y, incy)
palm_trigger.F90
#ifdef REAL_4
CALL saxpy(N, alpha, X, incx, Y, incy)
#else
CALL daxpy(N, alpha, X, incx, Y, incy)
#endif
Je vous rappelle que vous pouvez éditer le fichier
.ppl
à la main (après un peu d'expérience, ça
devient facile et rapide) et compiler les autres fichiers avec la
commande prepalm MOCAGE.ppl MOCAGE.pilN.B. PrePALM n'ouvre aucune fenêtre en mode compilation, mais il est écrit en langage Tcl/Tk et le shell associé a besoin d'accéder à un display X11 pour se lancer.
Compilation
On se positionne dans les répertoiresMOCAGE_COMPILE_ASSIMILATION/src_3DFGAT_SCHEMAPour le reste tout procède comme pour le modèle direct.
Regardons comment changent les fichiers
Make.commandsVoyons comment a changé par rapport au modèle direct le
Make.commands.pclinux
pour le schéma CARIOLLE#
# MOCAGE compilation suite
v.1.0.0
# Makefile include file
Make.commands
# for the CARIOLLE linear
chemical scheme
# running on a Linux PC under
PGI
#
# Scheme selection and specific
dependencies
#
SCHEME=CARIOLLE
SCHEME_DEPS=cariop_mod.F
#
# Machine dependent inlining
#
TCVQSAT_OBJ=tcvqsat.o
TCVQSAT_INLINE=
#
# CPP keys
#
CPPFLAGS=-DCARIOLLE -DLINO_3 -DPALM_RESEARCH -DUSE_BLAS -DMIDATM
-DNIV_60
#
# Compiler and linker commands
#
FC=mpif77
F90=$(FC)
LD=$(FC)
CC=
#
# Compilation flags as used in
the common Makefile
# (all of them should be
present even if empty or redefined)
#
# Specific include and lib paths
PALMHOME=/home/andrea/PALM/INSTALL/PALM_RESEARCH/MTSK_PAR/linux32r8lam
PALMINC=$(PALMHOME)/include
PALMLIB=$(PALMHOME)/lib
#
NOVINC=/home/andrea/USERS/andrea/WORK/NOVELTIS/V1.5/librairies/libmpr
NOVLIB=/home/andrea/USERS/andrea/WORK/NOVELTIS/V1.5/librairies/libsub
#NCDFINC=
#
# Generic fortran flags
FFLAGS=-r8 -pc 64 -byteswapio -O3 -Mdalign -Mextend -I$(PALMINC)
-I$(NOVINC)
#
# Specific flags for NEC high
vector optimisation
FFLAGSX=$(FFLAGS)
#
# Specific flags for NEC low
vector optimisation
FFLAGS0=$(FFLAGS)
#
# Specific flags for safe (e.g.
IEEE) compilation
FSAFEFLAGS=-r8 -pc 64 -byteswapio -O3 -Mdalign -Mextend
-I$(PALMINC) -I$(NOVINC) -Kieee
#
# Speficic fine optimisation
tuning flags
FVSAFEFLAGS=$(FFLAGS)
FSPHFLAGS=$(FFLAGS)
F_EXTRAFLAGS=
#
# OpenMP activitation flag at
compile time
F_OMPFLAGS=
C_OMPFLAGS=
#
# OpenMP activation flag at
link time
LD_OMPFLAGS=$(F_OMPFLAGS)
#
# Generic linker flags
LDFLAGS=$(FFLAGS) $(LD_OMPFLAGS) -L$(PALMLIB)
-L/usr/local/pgi/linux86/6.2/lib -L/home/andrea/USERS/andrea/XRD
-L/home/andrea/USERS/andrea/GRIBEX/gribex_000263 -L$(NOVLIB)
-L/home/andrea/USERS/andrea/WORK/NOVELTIS/V1.5/source/SPARSKIT
#
# Linked libraries
LIBS=-lxrd_jfe -lgribexR64 -lnetcdf -lpalm -lblas -lHnoveltis -lskit
On rappelle que la compilation peut être lancée par le script
jobMCGelf Par rapport à la compilation du modèle direct il faut changer la ligne
CASSIM=
en positionnant la variable à 1
Exécution
Rien de sorcier par rapport au modèle direct.C'est toujours le script
jobMCGelf qui pilote
l'exécution.La clé
CASSIM doit valoir 1.Les valeurs du nombre des threads OpenMP (cf. exécution du modèle direct) et de processeurs alloués doivent prendre en compte les ressources nécessaire à l'assimilation : nb processeurs = nb. de threads + 2.
Dans les lignes de configuration on trouve maintenant
TYPEOBS=MIPASavec une finalité exclusivement d'archivage des résultats.
Le choix de l'espèce contrôlée ne demande pas de recompiler le modèle ni de changer le schéma PrePALM. Il suffit de changer la ligne
ASSIMILATED_SPECIES="O_3"Tous le reste est identique au cas direct, le lancement aussi.
Bien sûr il y a bien plus de fichiers archivés dans
PATH_OUTPUT
:exp_description.lognamelist+datein+dateoutMOCAGE+datein+dateout.pplpalm_driver.outpalm_proc_001+datein+dateout.out
palm_proc_002+datein+dateout.outpalm_log+datein+dateout.outlbfgs+datein+dateout.outHMGLOB22+2003070103+I01.nc
HMGLOB22+2003070103+I02.nc
HMGLOB22+2003070106+I02.nc
HMGLOB22+2003070106+I03.nc
HMGLOB22+2003070109+I03.nc
HMGLOB22+2003070109+I04.nc
HMGLOB22+2003070112+I04.nc
HMGLOB22+2003070112+I05.nc
HMGLOB22+2003070115+I05.nc
HMGLOB22+2003070115+I06.nc
HMGLOB22+2003070118+I06.nc
HMGLOB22+2003070118+I07.nc
HMGLOB22+2003070121+I07.nc
HMGLOB22+2003070121+I08.nc
HMGLOB22+2003070124+I08.nc
HMGLOB22+2003070124+I09.nc
NCDF. Dans ce cas NCDF
= 6L'index
I## indique à quelle passage sur la
fenêtre le fichier a été produit. Dans ce cas le
paramètre ip_correctime est positionné
à ip_fenetre*0/3 et la fréquence des
sorties NetCDF à NCDF = 3A la première intégration le modèle a tourné de minuit à 3 heures. A 3 heures il écrit la sortie
2003070103+I01 qui contient donc l'ébauche à
3
heures.Après la première boucle d'assimilation on applique la correction à minuit et on intègre le modèle de minuit à 6 heures (on couvre la deuxième fenêtre).
Il écrit les sorties
2003070103+I02,
qui contient l'analyse à 3 heures, et 2003070106+I02
qui contient l'ébauche à 6 heures.Et ainsi de suite
2003070106+I03,
contient l'analyse à 6 heures, et 2003070109+I03
contient l'ébauche à 9 heures, etc.
HMGLOB22+2003070200NOUT
et en tout cas à la
date finale DATEOUT
(restart)HDIAGGLOB22+20030701+W01
HDIAGGLOB22+20030701+W02
HDIAGGLOB22+20030701+W03
HDIAGGLOB22+20030701+W04
HDIAGGLOB22+20030701+W05
HDIAGGLOB22+20030701+W06
HDIAGGLOB22+20030701+W07
HDIAGGLOB22+20030701+W08
HDIAGGLOB22+20030701+W09L'index
W## indique à quel passage ils ont
été calculés : le fichier W0N est
calculé avant assimilation sur la fenêtre N-1. Donc W01
contient la comparaison aux observations de l'ébauche sur les 3
premières heures. W02 contient la comparaison aux
observations de l'analyse entre minuit et 3 heures et de
l'ébauche entre 3 et 6 heures. W03 contient la comparaison aux observations de
l'analyse entre 3 et 6
heures et de
l'ébauche entre 6 et 9 heures et
ainsi de suite jusqu'à W09 qui contient la comparaison aux observations de
l'analyse sur la dernière fenêtreDXGLOB22+2003070100+O00.nc
DXGLOB22+2003070103+O003.nc
DXGLOB22+2003070106+O00.nc
DXGLOB22+2003070109+O00.nc
DXGLOB22+2003070112+O00.nc
DXGLOB22+2003070115+O00.nc
DXGLOB22+2003070118+O00.nc
DXGLOB22+2003070121+O00.ncHDOMD+datein+dateoutHDOMA+datein+dateout
HDOMF+datein+dateoutHSTAT+datein+dateout.ncEncore une fois faites attention aux vieux fichiers Arpège
HMGLOB22*
qui pourraient être restés dans le répertoire
d'exécution.Utilisation en mode chargeur NetCDF
Un cycle d'assimilation comporte au moins une intégration du modèle direct par fenêtre d'assimilation. Cette étape peut être coûteuse, en particulier avec les schémas complets ou la grille à haute résolution. Pour cette raison la version 4 offre la possibilité de relire les champs modèle issus d'une simulation précédente directement dans les sorties NetCDF. Deux applications importantes de cette option sont la comparaison à des nouveaux jeux d'observations indépendantes et la mise au point rapide de nouvelles options d'assimilation. Dans le premier cas, la comparaison aux observation est faite par l'opérateur H qui reçoit en entrée des champs modèle relus par une unité qui remplace le modèle MOCAGE tout en gardant la même interface PALM. L'unité chargeur récupère les sorties NetCDF à la fréquence spécifiée par la cléNCDF du script de lancement et les
interpole au pas de temps choisi comme "slot" d'assimilation
(normalement une heure). Si les sorties modèle ont
été stockées à la fréquence
correspondant au slot, on obtient le même résultat que par
intégration directe (avec la seule différence due
à la troncature en réel 4 octets au moment de
l'écriture dans les sorties NetCDF). Dans le deuxième
cas, l'assimilation se déroule comme avec le vrai modèle
avec la différence que sur la première fenêtre
l'intégration du modèle est remplacée par la
lecture de fichiers NetCDF et, à partir de la deuxième,
par la lecture des fichiers NetCDF auxquels on ajoute de façon
constante en temps, les incréments calculés sur les
fenêtres précédentesL'exécution est tout à fait semblable dans les deux cas : la modalité "comparaison aux observations" rentre dans le cas d'assimilation avec nombre d'itérations du minimiseur nul (
ip_niter=0
dans les constantes PrePALM).Dans la pratique, une répertoire
src_3DFGAT_NETCDF
a été crée dans MOCAGE_COMPILE_ASSIMILATION.
Il est tout à fait comparable aux autres répertoires
d'assimilation à la différence qu'il utilise le chargeur mocageNetCDF.F
(qui se trouve dans le répertoire MOCAGE_PALM)
à la place du modèle.Pour l'exécuter il faut indiquer dans le script de lancement
jobMCGelf
que le schéma chimique est CARIOLLE (N.B. c'est indépendant du
schéma chimique qui a produit les sorties NetCDF rélues).L'espèce chimique qui doit être rélue et comparée aux observations ou assimilée doit être indiquée dans la clé
ASSIMILATED_SPECIES
et, pour les nouvelles sorties, dans SPECIES
(après activation de CSHORT=1).La clé
NCDF doit indiquer la fréquence
à laquelle les sorties NetCDF sont disponibles.La clé
CNETCDF, si elle est positionnée
à 1, active la modalité par chargeur NetCDF.Le path
PATH_NC pointe vers le répertoire ou sont
stockées les sorties NetCDF : les noms des fichiers doivent
respecter la convention des sorties du modèle direct HMdomaine+date1+date2.nc (sans les
extensions des sorties d'assimilation +Ixx)Comment spécifier
les variances/covariances d'erreur d'ébauche
Avec l'approximation de la matrice B
par la solution d'une équation de diffusion
généralisée, les variances/covariances
d'erreur d'ébauche sont entièrement
spécifiées par le champs 3D des écarts type, le
champs des longueurs de portée horizontales et le champs des
longueurs de portée verticales.En l'absence d'informations statistiques sur les erreurs d'ébauche, une première approximation consiste à paramétrer les écarts type par une fraction de l'ébauche et à imposer des longueurs de corrélation horizontales et verticales homogènes et constantes. Ce choix a été le seul disponible jusqu'à la version 3 de la chaîne. Il est maintenant possible d'utiliser des variances et des longueurs de portée estimées qui permettent de prendre en compte la variabilité spatiale des statistiques d'erreur.
Voyons dans le détail par quelles constantes PrePALM et quels fichiers on spécifie ces quantités.
Pour ce qui est des variances, la constante
ip_Bvar_rep
indique le type d'approximation : ip_Bvar_rep=1 indique
que l'écart type est calculé comme une fraction de
l'ébauche. Dans ce cas, la constante ip_sigbck
indique le sigma (en %) pour le calcul de l'écart type =
concentration*ip_sigbck/100 ; ip_Bvar_rep=2
indique que la variance est considérée constante par
niveau mais différente pour chaque niveau. Dans ce cas, un
profil 1D des variances par niveau est lu dans la variable O_3_var_profile
du fichier BKG_ERR_STATS+200307.nc stocké dans le
répertoire indiqué par la clé PATH_STATS
(N.B. dans le cas
général O_3 sera remplacé par
l'espèce assimilée et 200307 par la date au
format yyyymm) ; ip_Bvar_rep=3
indique que la variance est estimée point par point et donc lue
comme un champ 3D dans la variable O_3_var_3d du fichier BKG_ERR_STATS+200307.nc.Pour ce qui est des corrélations horizontales, la constante
ip_Bhcor_rep
joue un rôle analogue, mais pour l'instant seul le choix ip_Bvar_rep=1
est implémenté : la longueur de portée horizontale
est homogène et constante. Elle est indiquée par la
constante ip_flsh (en degrés). (N.B. les calculs de diffusion pour
l'approximation de B se font
sur une grille de Gauss. La constante supplémentaire ip_scrip
indique par quel type d'interpolation on passe de la grille
modèle à la grille du vecteur de contrôle. Si ip_scrip=1
le package d'interpolation bilinéaire SCRIP est employé ;
si ip_scrip=0 le changement de grille se fait par
analyse/synthèse de Legendre à l'aide du package
SPHEREPACK)Pour ce qui est des corrélations verticales, la constante
ip_Bvcor_rep
permet de choisir l'approximation : ip_Bvcor_rep=1
indique que la longueur de portée verticale est homogène
et constante. Elle est indiquée par la constante ip_flsz
; ip_Bvcor_rep=2 indique que la longueur de portée
verticale varie selon la hauteur mais que le comportement se
répète à l'identique pour tous les points. Dans ce
cas, un profil 1D des longueurs de portée le long d'un profil
générique (indexé par niveau) est lu dans la
variable O_3_vcor_profile du fichier BKG_ERR_STATS+200307.nc
stocké dans le répertoire indiqué par la
clé PATH_STATS ; ip_Bvcor_rep=3
indique que la longueur de portée
verticale varie selon la hauteur de façon différente d'un
point à l'autre. Dans ce cas, un vrai champ 3D des longueurs de
portée est lu dans la variable O_3_vcor_3d du
fichier BKG_ERR_STATS+200307.nc.ip_vcor_1Dapprox=1
; si l'on veut prendre en compte la variabilité d'un profil
à l'autre (e.g. pour des études troposphériques
à haute résolution en présence d'un relief) il
faut positionner ip_vcor_1Dapprox=0 avec un
surcoût important en mémoire et en temps de calcul.Le fichier
BKG_ERR_STATS+yyyymm.nc
peut etre généré comme sortie d'un
traitement statistique (cf. les
travaux de S. Massart sur les statistiques d'ensemble) ou crée
à la main à partir d'un fichier ascii .cdl
par l'outil ncgen.
Deux exemples de fichiers .cdl sont donnés dans le
répertoire MOCAGE_PALM.Les deux traitements des
colonnes totales
L'assimilation des colonnes totales (ou partielles sans l'utilisation
explicite d'un averaging kernel) (type TOTCOL) peut se
faire de deux façon différentes, indiquées par la
constante PrePALM ip_tc_as_prof.Si
ip_tc_as_prof=0, la colonne est assimilé comme
un scalaire. L'opérateur d'observation H opère une sommation entre
deux limites en pression le long d'un profil avec des poids
proportionnels à l'épaisseur des couches. La
redistribution de l'incrément sur la verticale est
laissée à l'opérateur adjoint HT. Ce
choix est économique en termes de mémoire et de
coût de calcul, mais il est naturellement biaisé vers la
troposphère à cause de la plus grande contribution des
niveaux plus bas et plus espacés. Dans le cas d'assimilation de
l'ozone stratosphérique ceci comporte la tendance à faire
descendre excessivement le pic de concentration. Dans ce cas, le choix ip_tc_as_prof=1,
permet d'introduire un pseudo-profil observé
intermédiaire à ip_nivm1 niveaux, obtenu
à partir du profil d'ébauche multiplié fois le
rapport entre la colonne mesurée et la colonne
modélisée. Ceci permet de respecter la structure
verticale de l'ébauche, mais impose un surcoût en
mémoire dû au passage d'observations scalaires à
autant de profils à ip_nivm1 niveaux.Les erreurs les plus fréquentes
- L'intégration du modèle direct (ou
l'intégration du modèle dans le 3DFGAT) s'arrête
avec ce message
>> READING INITIAL FORCINGS : 2 DATES
> READING FORCING FILE (date A):FMGLOB22+2003070100
INVALID GEOMETRY FOR GRID (F1) : ABORT!
FORTRAN STOP
Il y a incohérence entre la résolution verticale sélectionnée à la compilation (clé CPP-DNIV_60) et la résolution verticale des fichiers de données (variableNIV=47dans le scriptjobMCGelf) - L'intégration du modèle direct (ou
l'intégration du modèle dans le 3DFGAT) s'arrête
avec ce message
*/*/* LFIOUV - Nom='HMGLOB22+2003070101'
***** LFIOUV - KREP= -9, KNUMER= 31, LDNOMM= T, CDSTTO='NEW', LDERFA= T, LDIMST= F, KNIMES= 0, KNBARP= 7 KNBARI= 0
***** LFIOUV - STATUS 'NEW' MAIS LE FICHIER EXISTE DEJA, UNITE 31 *****
ABOR2 CALLED
***** LFIOUV - STATUS 'NEW' MAIS LE FICHIER EXISTE DEJA, UNITE 31 *****
ABORT!! ***** LFIOUV - STATUS 'NEW' MAIS LE FICHIER EXISTE DEJA, UNITE 31 *****
Un vieux fichier ArpègeHMGLOB22+dateétait resté dans le répertoire d'exécution
- Le 3DFGAT s'arrête avec ce message
Warning: object size (7776000) exceeds per process memory (512000)dmem dump:
memory usage:
rank=0 freemem=512000 nobj=0
La taille du buffer de PALM est trop petite : il faut modifier la formule pour le calcul de la constante PrePALMip_buff_size.
- Le 3DFGAT s'arrête après la lecture des
forçages et à la fin du fichier
palm_proc_001.outil y a le message
>> PALM PARAMETERS DO NOT MATCH MOCAGE PARAMETERS
La période d'intégration du modèle est plus courte de la période d'assimilation : vérifiez que le paramètre PrePALMip_assimsizevaille au moinsip_nbfenet*ip_fenetre - Le 3DFGAT s'arrête avec l'un de ces messages (sortie
standard et fichier
palm_proc_002.out)
>> Number of levels 13 exceeds ip_nbvipresmax = 12
>> Number of profiles 501 exceeds ip_nobsmax = 500
>> Number of observed species 2 exceeds ip_nbconstsmax = 1
Les paramètres PrePALM de dimensionnement des observations (ip_nobsmax,ip_nbnivpresmax,ip_nbconstmax) sont insuffisants
Regardons les résultats
Nous avons choisi le format NetCDF pour les sorties car il s'agit d'un format auto descriptif très utilisé dans la communauté scientifique et géophysique en particulier (il a été retenu par l'IPCC, par exemple).Les noms des champs et leurs attributs suivent une convention très répandue dans la communauté climatique (convention CF). De nombreux logiciels de traitement de données et de visualisation savent tirer parti des informations codées selon cette norme. Nous verrons par la suite dans quel cas ceci nous sera utile.
La première commande à retenir est la commande
ncdump
qui permet de visualiser à l'écran le contenu d'un
fichier NetCDF.On l'utilise dans deux formes :
ncdump -h nomfichier.nc
pour visualiser les entêtes et la description des
variables contenues dans le fichieret
ncdump -v nomvariable
nomfichier.nc pour visualiser les valeurs d'une variable
contenue dans le fichierVoyons dans le détail le résultat de la commande
ncdump
-h HMGLOB22+2003070106.nc sur un fichier issu d'une simulation
directe avec schéma REPROBUS 60 niveaux.Dans ce cas, dans
jobMCGelfg nous avions demandé
de
sortir tous les niveaux (CLEVELS=0), de sortir les
colonnes totales aussi (CTOTCOL=1) et de ne sortir que
quelques espèces (CSHORT=1) et plus
précisément SPECIES="O_x O_3 CO".netcdf HMGLOB22+2003070106 {dimensions:
time = UNLIMITED ; // (1 currently)
lev = 60 ;
lat = 90 ;
lon = 180 ;
one = 1 ;N.B. l'attribut UNLIMITED permettra ensuite de concaténer plusieurs fichiers pour constituer une série temporelle
variables:
float time(time) ;
time:standard_name = "Time axis" ;
time:axis = "t" ;
time:units = "seconds since
2003-07-01 00:00:00" ;
time:time_origin = "2003-07-01
00:00:00" ;
time:calendar = "noleap" ;
float lev(lev) ;
lev:standard_name =
"atmosphere_hybrid_sigma_pressure_coordinate" ;
lev:positive = "down" ;
lev:formula_terms = "ap:
a_hybr_coord b: b_hybr_coord ps: air_pressure_at_surface" ;
lev:units = "lev" ;
float lat(lat) ;
lat:standard_name = "latitude" ;
lat:units = "degrees_north" ;
float lon(lon) ;
lon:standard_name = "longitude" ;
lon:units = "degrees_east" ;
float one(one) ;N.B. Le champ
time porte comme
attribut la date de référence et le type de calendrier
utilisé, pour pouvoir le positionner sur un axe absolu et le
champs lev porte comme attribut la formule pour obtenir la vraie
coordonnée pression float Dobson(one) ;
Dobson:long_name = "Dobson
constant for total column units conversion" ;one) Dobson contient le facteur de conversion
pour
l'unité de mesure des colonnes totales (cf plus bas) float air_pressure_at_surface(time, lat,
lon) ;
air_pressure_at_surface:standard_name = "air_pressure_at_surface" ;
air_pressure_at_surface:units =
"Pa" ;
float a_hybr_coord(lev) ;
a_hybr_coord:long_name = "a
coefficient of hybrid coordinate" ;
float b_hybr_coord(lev) ;
b_hybr_coord:long_name = "b
coefficient of hybrid coordinate" ;lev float air_temperature(time, lev, lat,
lon) ;
air_temperature:units = "K" ;
float x_wind(time, lev, lat, lon) ;
x_wind:units = "m s-1" ;
float y_wind(time, lev, lat, lon) ;
y_wind:units = "m s-1" ;
float w_wind(time, lev, lat, lon) ;
w_wind:units = "m
s-1" ; float pot_vorticity(time, lev, lat, lon) ;
pot_vorticity:units = "pvu" ;
float O_x(time, lev, lat, lon) ;
O_x:standard_name = "mole_fraction_of_O_x" ;
O_x:units = "ppv" ;
float O_x_tc(time, lat, lon) ;
O_x_tc:long_name = "Total column of O_x" ;
O_x_tc:units = "molec m-2" ;
float O_3(time, lev, lat, lon) ;
O_3:standard_name = "mole_fraction_of_O_3" ;
O_3:units = "ppv" ;
float O_3_tc(time, lat, lon) ;
O_3_tc:long_name = "Total column of O_3" ;
O_3_tc:units = "molec m-2" ;
float CO(time, lev, lat, lon) ;
CO:standard_name = "mole_fraction_of_CO" ;
CO:units = "ppv" ;
float CO_tc(time, lat, lon) ;
CO_tc:long_name = "Total column of CO" ;
CO_tc:units = "molec m-2" ;// global attributes:
:Conventions = "CF-1.0" ;
:source = "MOCAGE with chemical scheme REPROBUS" ;
:levels = 60 ;
:institution = "CNRM and CERFACS" ;Comme exemple d'utilisation de
ncdump -v regardons la
valeur de la constante Dobson
la commande ncdump -v Dobson HMGLOB22+2003070106donne comme résultat la même sortie de
ncdump -h
suivie dedata:
Dobson = 2.685e+20 ;
}On peut imposer la précision du résultat
ncdump -v Dobson -p 16 HMGLOB22+2003070106
donnedata:
Dobson = 2.685000006310853e+20 ;
}Voyons maintenant quels sont les outils qui accompagnent la maquette et qui permettent d'explorer les résultats.
Après avoir extrait le contenu de
MOCAGE_VISU.tgz
vous trouverez l'arborescence suivante :
NETCDF_AND_GRAPHICS/
README
NetCDF/
FERRET/
NetCDF/- Le répertoire
NetCDFcontient les deux scripts (un appelle l'autre en cascade) pour générer les méta fichiers (cf. plus bas) qui permettent d'analyser des séries temporelles à partir d'une collection de fichier NetCDF sur plusieurs dates. Il contient aussi la documentation sur l'interface Fortran/NetCDF et sur la partie de la convention CF qui concerne la chimie atmosphérique.
FERRET/- Le répertoire
FERRETcontient les scripts Ferret pour l'affichage des champs contenus dans les fichiers NetCDF avec le logiciel gratuit Ferret et quelques exemples de fichiers de palette pour le choix des couleurs.
Les fichiers Meta-NetCDF pour les séries temporelles
Le format d'archivage des sorties MOCAGE prévoit un fichier toutes lesNCDF heures. Les fichiers obtenus sont donc
suffisants pour réaliser des analyses ou des tracés
à un instant donné. Pour travailler sur des séries
temporelles on peut exploiter la capacité de certains logiciels,
dont Ferret, de lire des méta fichiers NetCDF (fichiers .mc)
et de rendre tout à fait transparent pour l'utilisateur
l'accès à la collection de fichiers. Il ne verra que des
champs dépendant du temps, dont l'axe temporelle couvre la
période des fichiers référencés par le
méta fichier.Puisque la syntaxe des fichiers
.mc est assez
délicate, dans le répertoire NetCDF vous
trouvez les deux scripts nc2mc et grgdif que
vous devez rendre accessibles à votre shell unix (les copier
dans $HOME/bin si vous en avez un, ou modifier votre
variable PATH).La commande
nc2mc -h vous montrera les options de nc2mc.Les options que nous utiliserons seront surtout
-n et -x
pour sélectionner les fichiers à prendre en compte.Dans l'exemple que nous avons pris en compte, nous disposons des fichiers
HMGLOB22+date.nc
à partir de HMGLOB22+2003070100.nc (puisque INICDF
était égal à 1) jusqu'à HMGLOB22+2003070200.nc,
toutes les 3 heures (NCDF=3). La commande
nc2mc -n HMGLOB22+200307 (négligez les
éventuelles requêtes de post-processing) vous produira le
fichier HMGLOB22+200307.mc qui nous servira par la suite.
Le nom du fichier .mc est automatiquement généré
à partir du préfixe de sélection, mais il
n'est pas important en soi. Nous pouvons donc lui attribue le nouveau
nom HMGLOB22+20030701.mc qui est plus parlant.La visualisation et le changement de coordonnées verticales avec FERRET
Ferret est un outils gratuit d'analyse de données et de visualisation développé au Pacific Marine Environmental Laboratory de la NOAA. Il tourne sous Unix, Linux, Windows et, en version beta, sous Mac OS X. Il lit plusieurs formats de fichiers, mais il a été optimisé pour lire du NetCDF. Il peut interagir avec les serveurs DODS/OPeNDAP pour accéder à des serveurs distants de données avec transfert minimal d'information.Ferret peut travailler en modalité ligne de commande (ce qui lui permet aussi d'être exécuté en batch pour la génération automatique de tracés opérationnels ou comme cgi-script en cas d'accès au données piloté par interface web) ou avec une interface graphique.
Je ne présenterai ici que les commandes à passer en mode ligne de commande, l'interface graphique étant encore plus intuitive.
Pour les commandes de base et l'utilisation de Ferret en général je renvoie à la page de documentation de Ferret.
Une séquence de commandes peut être enregistrée dans un fichier avec extension
.jnl. C'est la base des scripts
ferret qui sont appelés à partir de la ligne de commande
avec la syntaxego nom_du_script argument_1
argument_2 ...Dans le répertoire
FERRET vous trouvez des
scripts dont nous verrons l'utilité plus bas, outre à
deux fichiers .spk qui contiennent la définition de deux
palettes particulièrement bien adaptées à la
visualisation des champs chimiques. Avant de lancer Ferret, songez à positionner les variables d'environnement (chemins d'accès aux scripts, aux palettes et aux données d'orographie)
setenv FER_GO "$FER_GO $cequilfaut/NETCDF_AND_GRAPHICS/FERRET"et
setenv FER_PALETTE "$FER_PALETTE
$cequilfaut/NETCDF_AND_GRAPHICS/FERRET"et encore
setenv FER_DATA "$FER_DATA
$cequilfaut/NETCDF_AND_GRAPHICS/FERRET"Le prompt de Ferret est un sympathique
yes ?Pour lui indiquer de charger le contenu d'un fichier la commande est
USE
(majuscule ou minuscule ne font pas de différence)yes ? USE HMGLOB22+2003070106(l'extension
.nc est ajoutée par défaut, et
la touche Tab complète les noms de fichier)On peut charger plusieurs fichiers à la fois. Ils seront indexés avec un numéro de dataset (
d)
progressif.
Il sera toujours possible d'y faire référence par nom:d=1 sera équivalent à d=HMGLOB22+2003070106.ncLa commande
show data (tout raccourci non ambigu est
accepté, donc sh d convient aussi) montre le
contenu des datasets chargés. Nous retrouvons bien nos petitsyes? sh d
currently SET data sets:
1> ./HMGLOB22+2003070106.nc (default)
name
title
I
J
K L
DOBSON Dobson constant for total colum
1:1
...
... ...
AIR_PRESSURE_AT_SURFACE
1:180 1:90
... 1:1
A_HYBR_COORD
a coefficient of
hybrid coordin ...
...
1:60 ...
B_HYBR_COORD
b coefficient of
hybrid coordin ...
...
1:60 ...
AIR_TEMPERATURE
1:180 1:90
1:60 1:1
X_WIND
1:180 1:90
1:60 1:1
Y_WIND
1:180 1:90
1:60 1:1
O_X
1:180 1:90
1:60 1:1
O_X_TC Total column of
O_x
1:180 1:90
... 1:1
O_3
1:180 1:90
1:60 1:1
O_3_TC Total column of
O_3
1:180 1:90
... 1:1
CO
1:180 1:90
1:60 1:1
CO_TC Total column of
CO
1:180 1:90
... 1:1L'index
L fait référence au temps. A partir
de là vous pouvez explorer toutes les possibilités de
Ferret.Je montrerai seulement quelques exemples, avec la ligne qui a servi pour les générer. Ces plots serviront aussi de validation pour l'installation de la maquette (cliquez sur une petite image pour accéder à l'image en grandeur réelle).
La palette utilisée pour le tracé des champs s'appelle
caramel_bleu.
On l'active une fois pour toutes avec la commandepalette caramel_bleuou on peut la préciser dessin par dessin
fill/pal=caramel_bleu ....Pour le tracé de différences il peut être plus utile d'employer la palette
caramel_bleu_ctr, où la
valeur 0 correspond au blanc.Voyons donc pour le schéma REPROBUS la colonne totale d'O_x en Dobson
fill/title="Colonne totale O_x (DU)" O_X_TC/Dobson
contour/ov/title="O_x" O_X_TC/Dobson
go land 2Voilà qu'une différence entre la colonne totale de O_x et de O_3 est vite réalisée
fill/title="Diff Col tot O_x-O_3 (DU)" (O_X_TC -
O_3_TC)/Dobson
contour/ov/title=" " (O_X_TC - O_3_TC)/Dobson
go land 2
Pour obtenir plus de détail sur les régions
où la
différence est significative nous pouvons utiliser la palette
avec du blanc au centre et recentrer les niveaux autour de 0fill/title="Diff Col tot O_x-O_3
(DU)"/pal=caramel_bleu_ctr/levels=(-1.1,1.1,.1) (O_X_TC - O_3_TC)/Dobson
contour/ov/title=" " (O_X_TC - O_3_TC)/Dobson
go land 4Une moyenne zonale n'est pas difficile non plus
fill/title="O_x (ppb)" O_x[x=@ave]*1.e6
contour/ov O_x[x=@ave]*1.e6Si nous voulons représenter le même champ en concentration, ou avec des coordonnées verticales différentes, nous utilisons le script vert_coord.jnl qui prend en argument le nom de la variable à transformer.
go vert_coord O_3
donnera comme résultat
Vertical coordinate conversion for variable O_3
In datafile HMGLOB22+2003070106 d=1
Temperature from file HMGLOB22+2003070106
Orography from file OROGRAPHY_GLOB22.nc
RESULT variables
1] O_3_p = O_3 on pressure vertical coordinate
2] O_3_th = O_3 on potential temperature vertical coordinate
3] O_3_km = O_3 on height vertical coordinate
4] O_3_c = O_3 concentration
5] O_3_c_p = O_3 concentration on pressure vertical coordinate
6] O_3_c_th = O_3 concentration on potential temperature vertical
coordinate
7] O_3_c_km = O_3 concentration on height vertical coordinate
8] O_3_pp = O_3 partial pressure
9] O_3_pp_p = O_3 partial pressure on pressure
vertical
coordinate
10] O_3_pp_th = O_3 partial pressure on potential
temperature vertical coordinate
11] O_3_cp_km = O_3 partial pressure on
height vertical coordinate
WORK ARRAYS still defined
a] air_pressure = 3D air pressure field
b] air_density = 3D air density field
c] air_theta = 3D air potential temperature field
d] height = 3D height of the model grid points
Il suffira donc de tracer
fill O_3_c[x=@ave]Regardons les images que l'on peut produire à partir des trois autres coordonnées verticales : pression, température potentielle et altitude.
La moyenne zonale n'est pas très significative sur un plot en pression avec axes linéaires. Pour cette raison nous avons codé des scripts pour la représentation avec axe vertical en logarithme pression. Donc, si nous utilisons le script lat_logp.jnl (tracés sur un repère latitude / log de la pression), dont les premières lignes de commentaires indiquent les options disponibles, nous obtenons
go lat_logp O_3_p
go lat_logp O_3_p "" "" "" "" "" "" contour/ovUn outil analogue
lon_logp.jnl permet de tracer des
coupes longitude vs. log(p)
go lon_logp O_3_p "" "y=45N"
go lon_logp O_3_p "" "y=45N" "" "" "" "" contour/ov
fill O_3_km[x=@ave]
contour/ov O_3_km[x=@ave]
.mc
dépendants du temps On les charge avec la commande
set data set data HMGLOB22+20030701.mcSi vous regardez les données auxquelles Ferret peux maintenant accéder (
sh d) vous verrez que
l'axe du temps couvre 9 dates, du 01-JUL-2003:00:00 au 02-JUL-2003:00:00
par pas de 3 heures.Nous pouvons donc tracer des Hoevmoellers, par exemple des moyennes horizontales globales
fill/title="O_x (ppb)" O_x[x=@ave,y=@ave]*1.e6
contour/title="O_x"/ov O_x[x=@ave,y=@ave]*1.e6Pour obtenir le tracé correspondant avec coordonnée verticale pression nous utilisons la procédure vert_coord.jnl, comme pour les tracés à une date fixe. Pour avoir un axe vertical logarithmique, il faut ensuite appeler le script
time_logp
go vert_coord O_X
go time_logp O_X_p
go time_logp O_X_p "" "" "" "" "" "" contour/ov
O_x
ajouté à 9 heures (avec insertion de la correction au
début de la fenêtre d'assimilation), donc de
l'incrément produit au cours de la quatrième
fenêtre d'assimilation (de 9 heures à midi).Il se trouve dans le fichier
DXGLOB22+2003070109+O00.ncNous en traçons la moyenne zonale
fill/title="Moy zon incr (ppb)" incr[x=@ave]*1.e6et le champ 2D en correspondance du niveau 13
fill/title="Increment (ppb)" incr[k=13]*1.e6Pour dessiner, enfin, l'incrément à un niveau de pression constant, on n'a pas besoin d'interpoler le champ sur la coordonnée verticale pression.
Il suffit de reconstruire le champs de pression 3D à partir des coefficients de la coordonnée sigma et de la pression de surface et d'utiliser la fonction de localisation
@weq de FERRET.
Par exemple, pour tracer l'incrément d'analyse à une
pression donnée (10hPa dans l'exemple) il suffira de donner
à FERRET les commandes :use DXGLOB22+2003070109+O00.nc
let air_pressure=A_HYBR_COORD+B_HYBR_COORD*AIR_PRESSURE_AT_SURFACE
let kern=O_3*air_pressure[z=@weq:1000]*1.e6
let hsect=kern[k=@sum]
palette caramel_bleu_ctr
fill/lev=(-2,2,.2)/title="Increment at 10hPa" hsect
go land
L'analyse des performances avec PrePALM
La dernière image n'est pas issue des calculs de MOCAGE mais est un exemple de l'analyseur de performances de PALM.Une telle image vous permet de vous assurer qu'il n y a pas de goulot d'étranglement de votre application, que la charge est relativement bien repartie entre les deux processeurs de travail et, surtout qu'il n'y a pas eu de fuite de mémoire au niveau du buffer, ni qu'il est surdimensionné. On génère cette image en chargeant le fichier de statistiques palm_log.out à partir du menu Analyse run r en suivant d'abord Load file et ensuite Performances analyser.
Pour plus de détail sur la lecture de ce graphique, nous renvoyons à la documentation de PALM.
Les diagnostiques
d'assimilation automatiques
Après une expérience d'assimilation il est pratique de
sortir des tracés standard, de façon à pouvoir
comparer les résultats entre différentes
expériences. Pour cette raison, nous avons introduit deux
nouvelles procédures FERRET pour tracer les statistiques
d'assimilation. La première, dailystats.jnl, produit des
graphiques détaillés, fenêtre par fenêtre et
selon le type d'instrument, des écarts modèle/observation
avant et après assimilation. La deuxième,
thermostats.jnl, fournit des statistiques plus adaptées à
des analyses plus longues, couvrant un mois au maximum. Pour la
correcte génération de fichiers mufti-images, l'outil
gifsicle doit être installé et accessible. Il appartient
au package CDAT, ou il est distribué seul.Une variante de ces procédures, adaptée au cas des modèles avec coordonnée verticale en pression pure (p.e. MSDOL), se trouve dans le répertoire
MB). Dans ce
cas, les scripts s'appellent dailystatsmb.jnl et monthlystatsmb.jnl.Le script dailystats
doit être
utilisé à partir d'un répertoire d'archivage des
résultats d'assimilation (il utilise les différents
fichiers NetCDF produits par la chaîne). Il reçoit en argument la date du jour au format yyyymmdd.
Un deuxième argument optionnel indique le nombre de sections horizontales pour les instruments de type profil (cf. plus bas). Par défaut les niveaux des sections sont 10 et partagent (au sens logarithmique) l'intervalle entre la plus haute et la plus basse des observations. Un argument entier indiquer un nombre de niveaux différent pour partager la même intervalle. Si l'argument est 0, les niveaux ne sont pas calculés, mais lus dans le fichier
def_sections.jnl
qui doit se trouver soit avec les données, soit avec les autres
scripts FERRET. Un fichier d'exemple est distribué avec les
autres scripts. Notez que dans ce cas vous pouvez de façon
optionnelle, préciser les valeurs des isolignes niveau par
niveau. Ceci est utile, par exemple, pour réaliser des
animations.Rappelons que ferret peut être lancé en modalité batch, sans affichage :
ferret -gif -script dailystats 20030701Voyons ce qu'il produit pour une date donnée, instrument par instrument :
Pour une quantité scalaire (
TOTCOL ou INTQTY) :- date
+instrn+totcol+obs_nr.gif - Le nombre d'observations, slot par slot
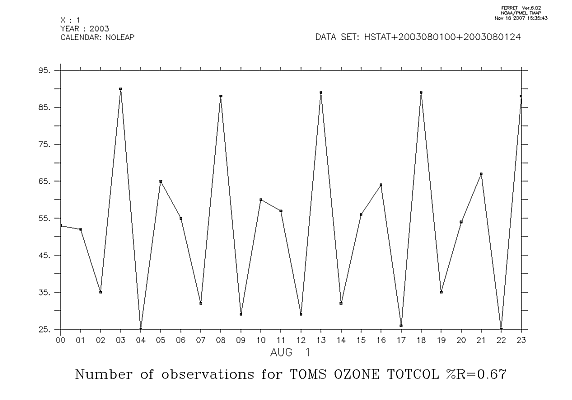
- date
+instrn+totcol+fct.gif - Fichier multi-image (une par fenêtre) où les valeurs
des données sont superposées au champs scalaire issu de
la prévision. Les fichiers date
+instrn+totcol+ana.gifet date+instrn+totcol+dry.gifcontiennent les mêmes images pour l'analyse et pour le dry run 
- date
+instrn+totcol+omf.gif - L'écart en % entre observations et prévision. Les
fichiers date
+instrn+totcol+oma.gifet date+instrn+totcol+omd.gifcontiennent les mêmes images pour l'analyse et pour le dry run 
- date
+instrn+totcol+inc@xave.gif - Fichier multi-image (une par fenêtre) avec les moyennes
zonales des incréments d'analyse. Le fichier date
+instrn+totcol+inc@yave.gifcontient les moyennes longitudinales 
Pour un profil :
- date
+instrn+profile+obs_nr.gif - Le nombre d'observations, slot par slot
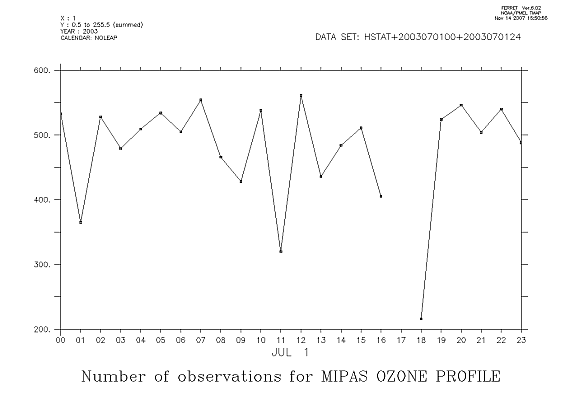
- date
+instrn+profile+fct@yyyPa.gif - Fichier multi-image (une par fenêtre) où les valeurs
des données sont superposées à la section à
yyy Pa du champs de la
prévision. Les fichiers date
+instrn+profile+ana@yyyPa.gifet date+instrn+profile+dry@yyyPa.gifcontiennent les mêmes images pour l'analyse et pour le dry run 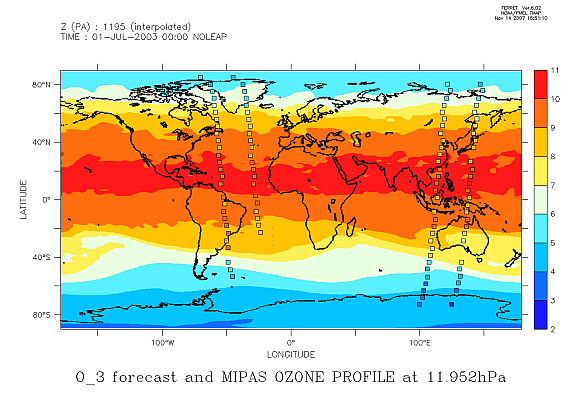
- date
+instrn+profile+inc@yyyPa.gif - Fichier multi-image (une par fenêtre) avec les sections à yyy Pa de l'incrément d'assimilation et la superposition des observations qui ont pu affecter le niveau à yyy Pa.
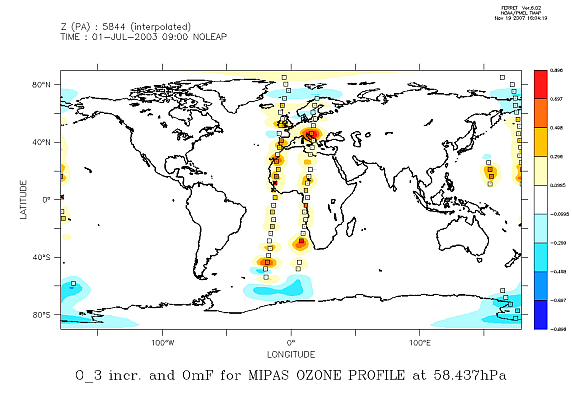
- Les observations rejetées à cause du dépassement du seuil sur le misfit sont barrées d'une croix
- date
+instrn+profile+OmDFA-Global.gif - Fichier multi-image (une par fenêtre) avec les moyennes
scalaires de l'écart aux observations niveau par niveau pour
prévision, analyse et dry run. Le moyennes sont calculées
sur la totalité du globe ou par tranche de latitude (fichiers date
+instrn+profile+OmDFA-90S60S.gif, date+instrn+profile+OmDFA-30S30N.gifet date+instrn+profile+OmDFA-60N90N.gif) 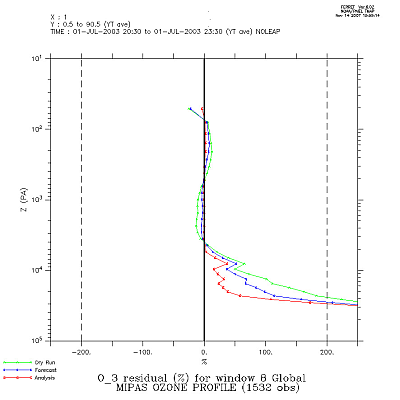
Le script monthlystats aussi doit être utilisé à partir d'un répertoire d'archivage des résultats d'assimilation.
Il reçoit en argument le mois de l'analyse au format yyyymm. Le script peut être lancé même si l'analyse pour le mois n'est pas complète. Les statistiques seront calculées seulement pour les jours analysés.
Il produit pour un mois donné :
chi2+date.gif- Le test chi 2 sur toutes les fenêtres d'assimilation, avec moyenne glissante et moyenne sur le mois
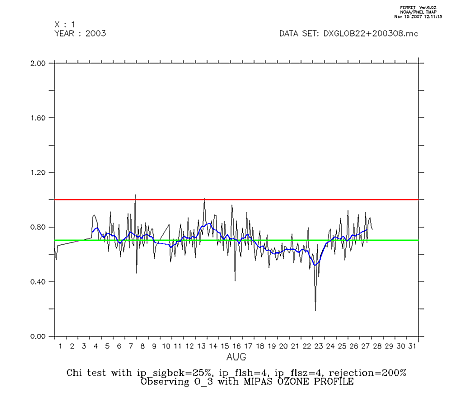
average_espece_incr+date.gif- La moyenne zonale et sur le mois de l'incrément d'assimilation
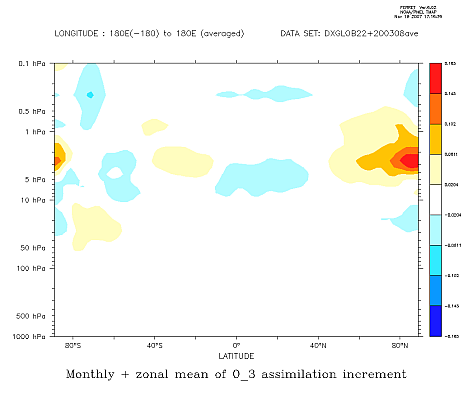
- date
+OmDFA-Global.gif - Les moyennes scalaires de l'écart aux observations niveau par niveau pour prévision, analyse et dry run. Le moyennes sont calculées sur la totalité du globe ou par tranche de latitude
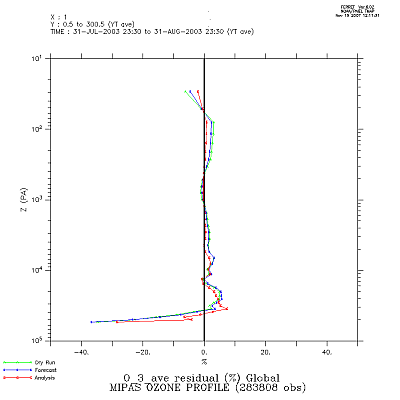
Des idées pour le futur
Nouvelles formulations des corrélations de l'erreur d'ébauche
La matrice de variance/covariance des erreurs d'ébauche n'est pas explicitement représentée dans le 3DFGAT. À sa place nous utilisons une approximation du produit de la racine carrée (au sens matriciel, cf. la section sur l'algorithme d'assimilation) de la matrice fois un vecteur. Cette approximation est basée sur la solution d'une équation de diffusion. Dans la version V4 on utilise un schéma spectral pour sa solution 2D sur la sphère. Cette approximation est l'objet d'une intense activité de recherche et elle est destinée à évoluer rapidement. En particulier nous introduirons des corrélations anisotropes et une estimation adaptative des coefficients multiplicatifs pour les variances, basée sur les travaux de G. Desroziers. Une autre piste à l'étude est la reformulation du problème de minimisation avec utilisation directe des coefficients spectraux comme variable de contrôle.Contrôler plusieurs espèces à la fois
La dernière évolution que l'on puisse envisager consiste en augmenter la taille de l'espace de contrôle. Ainsi pourrait-on corriger plusieurs espèces à la fois. Même dans le cas le plus simple, dans lequel on assimile des observations indépendantes pour chacune des espèces contrôlées (assimilation monovariée), cette étape demande une profonde refonte du 3DFGAT, car la forme de repérage des espèces contrôlées à l'intérieur du vecteur des espèces pronostiques ne pourra plus se limiter à un seul index. De plus, les tailles du vecteur de contrôle et de tous les espaces qui en dépendent devront être paramétrées dans les cartes d'identité des unités. Encore plus compliqué est le cas avec vraie assimilation multivariée, dans laquelle les corrections sur les différentes espèces ne sont pas indépendantes, mais liées par les blocs hors-diagonaux de la matrice d'erreur d'ébauche B.On peut imaginer qu'une modification d'une telle ampleur sera réservée pour une phase ultérieure d'évolution du système d'assimilation.